专题:RNA-Seq 上游分析学习
本文总阅读量 次
转录组测序的研究对象为特定细胞在某一功能状态下所能转录出来的所有 RNA 的总和,包括 mRNA 和非编码 RNA,相对于传统的芯片杂交平台,转录组测序无需预先针对已知序列设计探针,即可对任意物种的整体转录活动进行检测,提供更准确的数字化信号,更高的检测通量以及更广泛的检测范围,是目前深入研究转录组复杂性的强大工具,基于高通量测序平台的转录组测序技术能够全面获得物种特定组织或器官的转录本信息,从而进行基因表达水平研究、新转录本发现研究、转录本结构变异研究等。
RNA-seq 概述
RNA-seq 是研究转录组应用最广泛,也是最重要的技术之一,RNA-seq 分析内容包括序列比对、转录本拼接、表达定量、差异分析、融合基因检测、可变剪接、RNA 编辑和突变检测等,具体流程和常用工具如下图所示,通常的分析不一定需要走完全部流程,按需进行,某些步骤可以跳过、简化等。

RNA-seq 中最常用的分析方法就是找出差异表达基因 (Differential gene expression, DEG),在实验室中,标准流程就分为三步:
- step1: 构建测序文库,包括提取 RNA, 富集 mRNA 或清除核糖体 RNA, 合成 cDNA, 加上接头
- step2: 在高通量测序平台(通常为 Illumina) 上对文库进行测序,每个样本的测序深度为 10-30M 读长
- step3: 数据分析,具体而言:对测序得到的读长进行比对或组装到转录本上;对覆盖到每个基因区域的读长进行计数;根据统计模型鉴定不同样本间差异表达的基因,(这种分析过程是比较传统的方法)
版本信息
- 成都理工大学超算平台 Red Hat 4.8.5-36
- conda 4.10.3
- 测序数据来自 NCBI ACC=SRR25909836
- 参考基因组和注释文件来自 Ensembl Plants
- 物种:Oryza sativa Japonica
一些名词解释
- adapter:接头,为一段已知的短核苷酸序列,用于链接未知的目标测序片段
- index:几个碱基组成的寡核苷酸链,用于在混合测序时,区分不同样本
- insert:待测序的目标序列,位于两个 adapter 之间
- reads:在测序过程中,从样本中识别出来的 DNA 或 RNA 序列
- Transcriptome: 包含所有 RNA 分子的集合,可以用来量化每个基因的表达水平
- Expression levels 或 Coverage: 表示特定基因的 RNA 数量,通常用来衡量基因的表达水平
- Differential expression analysis:用来比较不同样本或不同处理组之间的基因表达差异
- Gene annotation:一个包含有关每个基因的基本信息的数据库,如其位置,功能等
- Splice variants:一个基因能够通过不同的剪接方式生成不同的 RNA,这些不同的 RNA 就叫做剪接异构体
- FPKM (Fragments Per Kilobase of transcript per Million mapped reads) 和 TPM (Transcripts Per Kilobase Million):用作基因表达水平的衡量标准
- Quality control:确保数据质量,清理模糊不清或者质量低下的 reads
- Genome:一个生物体的所有遗传信息
大致流程

软件安装
只需要使用 conda 就可以安装所有需要的软件,主要使用的软件有以下一些
- sra-tools:快速下载 NCBI SRA 数据
- fastQc:测序数据质量检测与控制
- multiqc:合并质量检测报告
- trimmomatic: 过滤低质量序列
- hisat2:转录组数据的比对
- samtools:对 hisat2 比对的结果进行排序和压缩
- featureCounts:对基因的信息进行计数统计
先创建 conda 虚拟环境,安装所需要的软件,可以自行手动安装,也可以直接导入我的 conda 环境:
git clone https://github.com/yuanj82/NGS-analysis.git
cd NGS-analysis/environment
cp .condarc ~/
conda env create --file rna-seq-env.yml # RNA-seq
创建完成后激活环境就可以使用了:
conda activate rna-seq
如果想自己安装也可以,用下列命令就可以:
conda install bioconda::sra-tools
conda install bioconda::fastqc
conda install bioconda::trimmomatic
conda install bioconda::samtools
conda install bioconda::hisat2
conda install bioconda::subread
conda install bioconda::multiqc
数据获取与预处理
测序数据下载
先在 NCBI 的 SRA 数据库搜索感兴趣的物种

选择符合自己要求的文章,找到下面 Runs 这里,点击 SRR 开头的编号
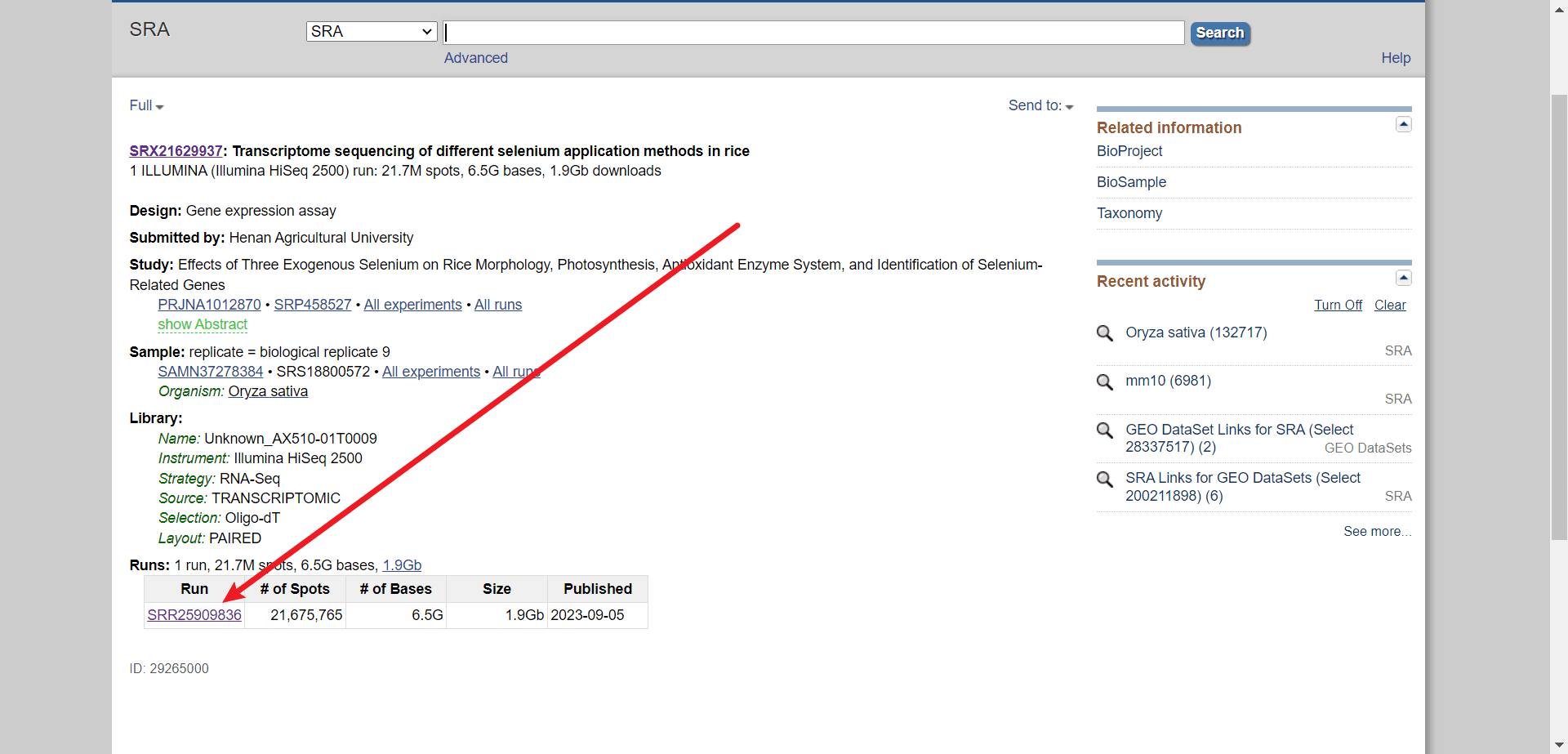
查看数据是否符合要求
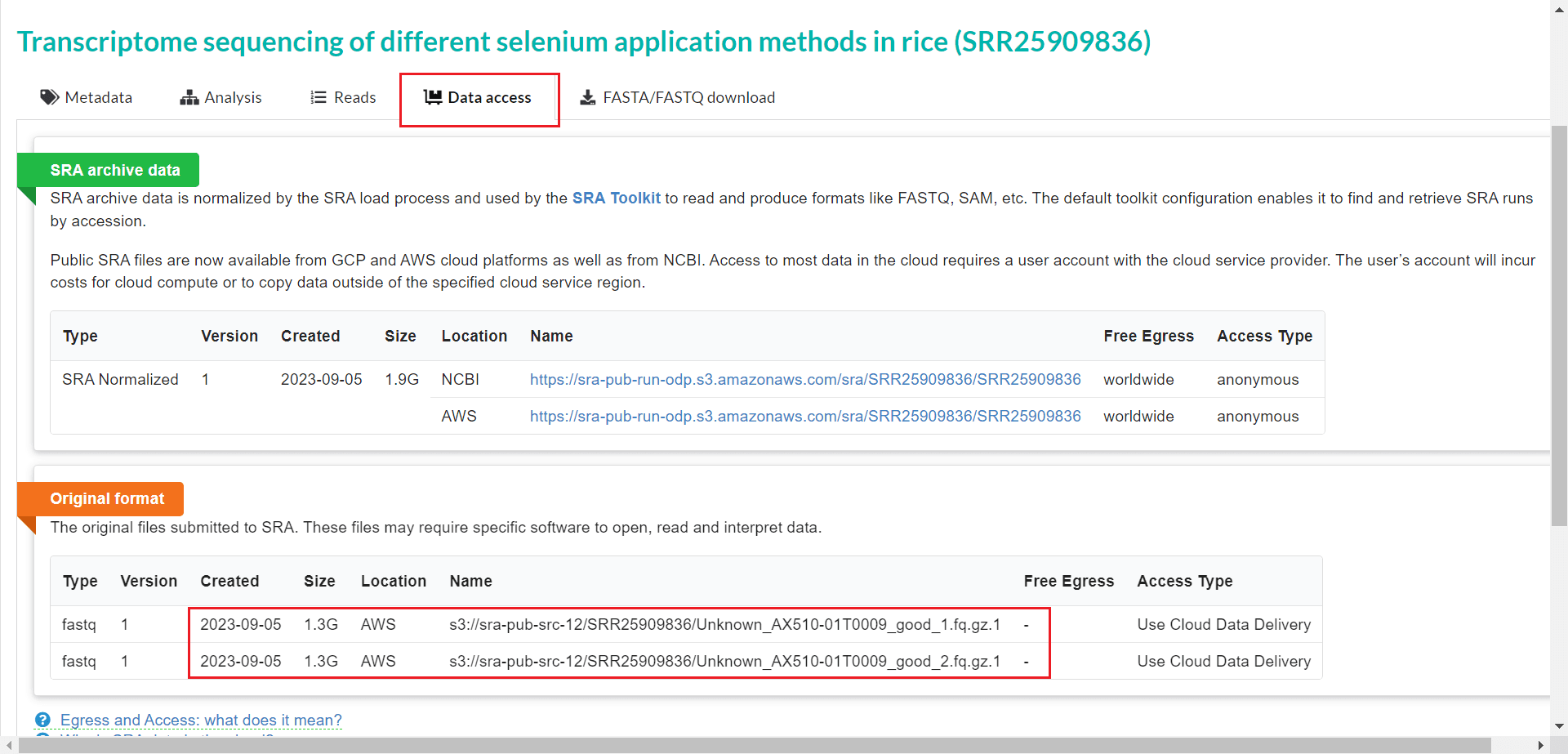
文件是***_1.fq.gz、***_2.fq.gz 这种是双端测序数据,我们需要这种双端测序的数据来进行 RNA-seq 分析
如果数据是双端测序的,那么就复制 SRR 编号,使用 sratools 下载,例如:
prefetch SRR8956151
批量下载需要先建立一个 txt 文件,将 SRR 编号写进去,例如:
SRR5830630
SRR5830631
SRR5830632
SRR5830633
SRR5830634
然后使用下面的命令下载
prefetch --option-file SRR_Acc_List.txt
由于数据比较大,可以使用 nohup 命令挂在后台下载
nohup prefetch -O . $(<SRR_Acc_List.txt) &
刚刚下载好的数据是 sra 格式的,使用 sratools 将其拆分
fastq-dump --gzip --split-3 SRR25909836.sra
- –gzip 是将拆分的 fastq 文件压缩归档为 gz 格式
- –split-3 是将文件拆分为正向序列和逆向序列
如果数据比较多,就写一个 bash 脚本
#!/bin/bash
mkdir SRR
cat SRR_Acc_List.txt | while read id; do mv -f ${id}/${id}.sra ./SRR; done
cd SRR
for i in *sra
do
fastq-dump --gzip --split-3 ${i} -f ../fastqgz
done
参考基因组及注释文件
植物的我一般在 Ensembl Plants 下载,用 wget 或 curl 都可以,内存不大
wget https://ftp.ensemblgenomes.ebi.ac.uk/pub/plants/release-57/fasta/oryza_sativa/dna/Oryza_sativa.IRGSP-1.0.dna.toplevel.fa.gz
wget https://ftp.ensemblgenomes.ebi.ac.uk/pub/plants/release-57/gff3/oryza_sativa/Oryza_sativa.IRGSP-1.0.57.gff3.gz
然后解压
gzip -d Oryza_sativa.IRGSP-1.0.57.gff3.gz
gzip -d Oryza_sativa.IRGSP-1.0.dna.toplevel.fa.gz
为了方便,我把两个文件分别重命名为 oryza_sativa.fa 和 oryza_sativa.gff3
mv Oryza_sativa.IRGSP-1.0.dna.toplevel.fa oryza_sativa.fa
mv Oryza_sativa.IRGSP-1.0.57.gff3 oryza_sativa.gff3
FastQc 质控
常用参数
fastqc [-o output dir] [--(no)extract] [-f fastq|bam|sam] [-c contaminant file] seqfile1 .. seqfileN
- -o –outdir:FastQC 生成的报告文件的储存路径,生成的报告的文件名是根据输入来定的
- –extract:生成的报告默认会打包成 1 个压缩文件,使用这个参数是让程序不打包
- -t –threads:选择程序运行的线程数,每个线程会占用 250MB 内存,越多越快咯
- -c –contaminants:污染物选项,输入的是一个文件,格式是 Name [Tab] Sequence,里面是可能的污染序列,如果有这个选项,FastQC 会在计算时候评估污染的情况,并在统计的时候进行分析,一般用不到
- -a –adapters:也是输入一个文件,文件的格式 Name [Tab] Sequence,储存的是测序的 adpater 序列信息,如果不输入,目前版本的 FastQC 就按照通用引物来评估序列时候有 adapter 的残留
- -q –quiet:安静运行模式,一般不选这个选项的时候,程序会实时报告运行的状况
进行质量检测
这里我们使用 fastqc [文件名] 即可
fastqc SRR25909836_1.fastq.gz
当然,数据比较多的时候还是挂在后台批处理然后等着就行
nohup fastqc SRR*.fastq.gz &
multiqc ./fastqc_report # 将多个质量检测报告合并
程序运行完成后会输出一堆 html 文件和 zip 压缩包,html 是网页版报告,zip 是本地宝报告,下载到本地用浏览器打开就可以看到质量检测报告了

左侧 Summary 部分就是整个报告的目录,整个报告分成若干个部分
- 合格:绿色的√
- 警告:黄色的!
- 不合格:红色的×
我们一般比较关心的是下面几个部分
- Basic Statistics: 对数据量的概览
- Per base sequence quality:reads 每个位置测序质量最直接的展示
- Per sequence quality scores:总体 reads 测序质量趋势
- Per base sequence content: ATGC 含量估计测序是否存在偏差
- Sequence Duplication Levels:影响测序的因素太多,查看是否存在污染,数据处理时是否需要去冗余;现在数据量都可以满足需求,因此前期数据处理时,尽量高标准,严格质控
Basic Statistics

每个位置的碱基的测序质量
- Encoding:指测序平台的版本和相应的编码版本号
- Total Sequences:总的 reads 数
- Sequence length:测序的长度
- %GC:是我们需要重点关注的一个指标,这个值表示的是整体序列中的 GC 含量,这个数值一般是物种特异的,比如人类细胞就是 42%左右,如果测序原始数据的 GC 含量远远偏离这个比例,说明测序数据存在一定偏好性,如果直接用测序数据,会影响后续的 CNV 和变异检测的分析
Per base sequence quality

- 横轴代表位置(第 1 到 150 个碱基)
- 纵轴代表 quality
- 红色表示中位数
- 黄色是 25%-75%区间
- 触须是 10%-90%区间
- 蓝线是平均数
- Warning,如果任何碱基质量低于 10, 或者是任何中位数低于 25
- Failure,如果任何碱基质量低于 5, 或者是任何中位数低于 20
一般要求此图中,所有位置的 10%分位数大于 20,否则切除 20 以下的碱基,从而保证后续分析的正确性
Per tile sequence quality
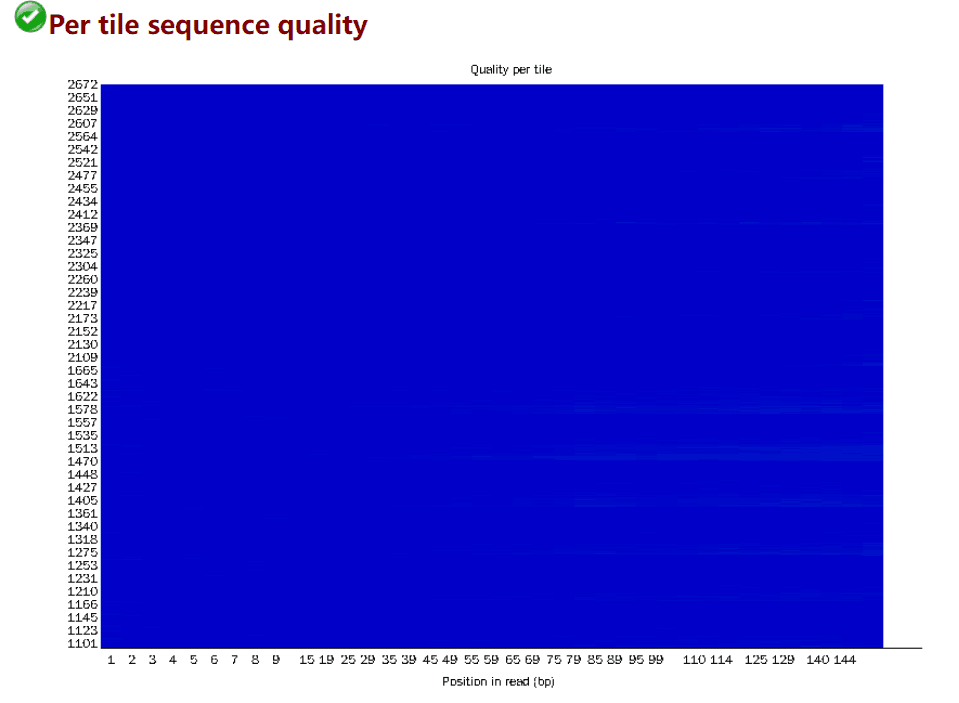
每个 tile 测序的测序质量
- tile:每一次测序荧光扫描的最小单位
- 横轴代表 101 个碱基的位置
- 纵轴是 tail 的 Index 编号
检查 reads 中每一个碱基位置在不同的测序小孔之间的偏离度,蓝色表示低于平均偏离度,偏离度小,质量好;越红表示偏离平均质量越多,质量也越差,如果出现质量问题可能是短暂的,如有气泡产生,也可能是长期的,如在某一小孔中存在残骸,问题不大,可以看到我的这个数据几乎没有瑕疵
Per sequence quality scores
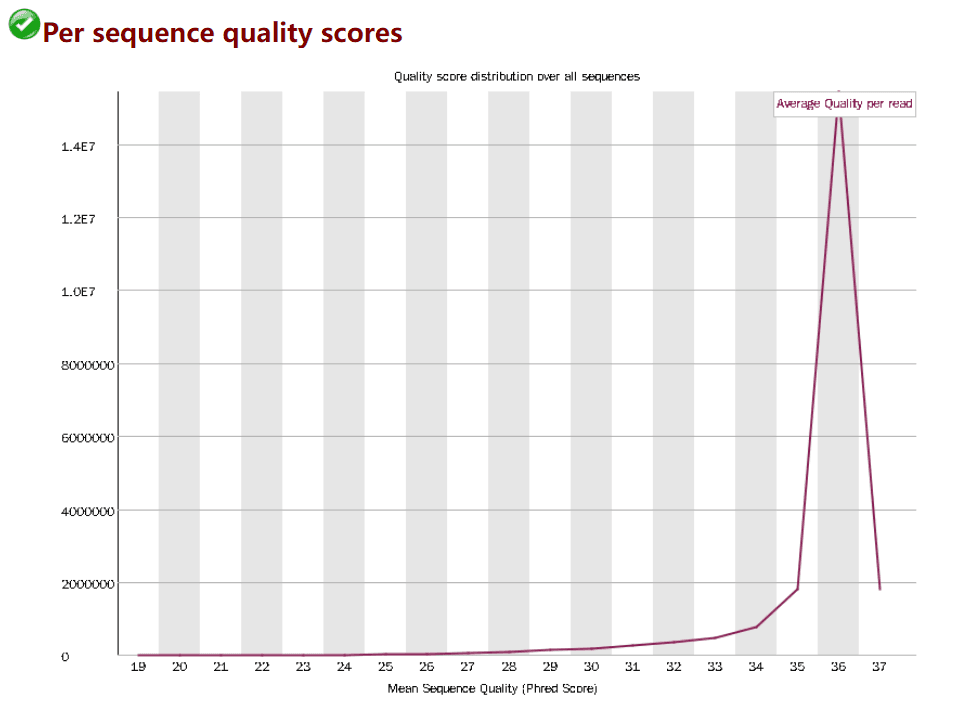
每条序列质量得分的分布情况
- 横轴是平均质量得分
- 纵轴收到该平均质量得分的 reads 数目
- 当测序质量峰值小于 27(错误率 0.2%)时报"WARN"
- 当峰值小于 20(错误率 1%)时报"FAIL"
假如我测的 1 条序列长度为 101bp,那么这 101 个位置每个位置 Q 值的平均值就是这条 reads 的质量值,我这个数据的质量不错,reads 大都集中在高分上,纵轴数值越大,该序列测序错误的可能就越小
Per base sequence content
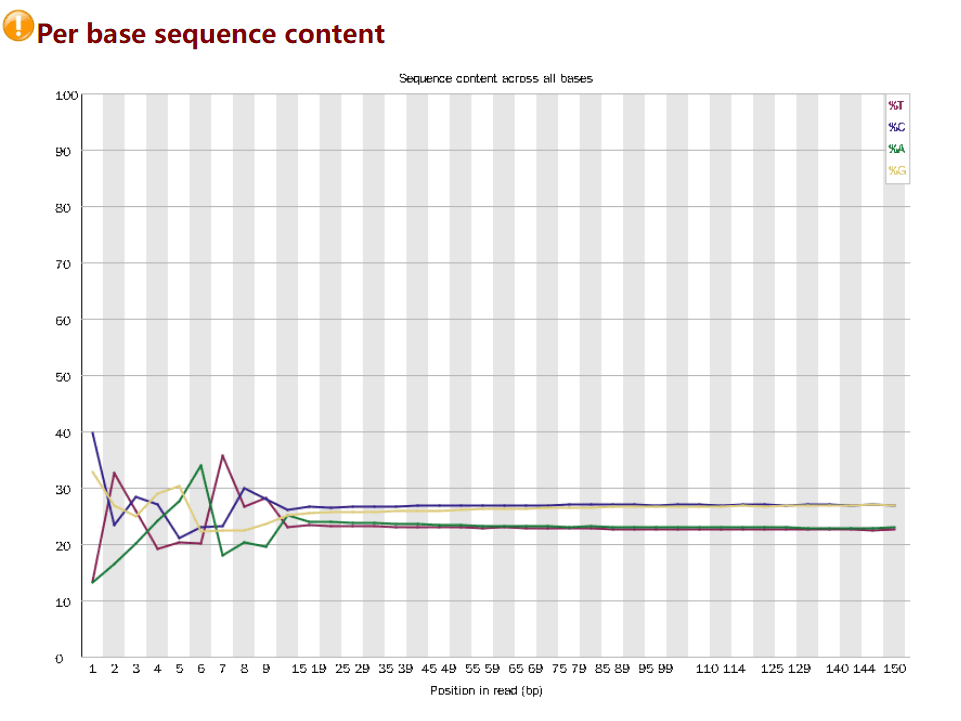
统计 reads 每个位置 ATCG 四种碱基的分布
- 横轴是 1 - 101 bp;纵轴是百分比
- 图中四条线代表 A T C G 在每个位置平均含量
- 理论上来说,A 和 T 应该相等,G 和 C 应该相等,但是一般测序的时候,刚开始测序仪状态不稳定,很可能出现上图的情况,像这种情况,即使测序的得分很高,也需要 cut 开始部分的序列信息
Per sequence GC content

序列平均 GC 含量分布
- 横轴是百分比
- 纵轴是每条序列 GC 含量对应的数量
- 蓝色的线是程序根据经验分布给出的理论值
- 红色是真实值
蓝红色线应该比较接近才比较好,当红色的线出现双峰,很有可能是混入了其他物种的 DNA 序列,比如我这张图
Per base N content
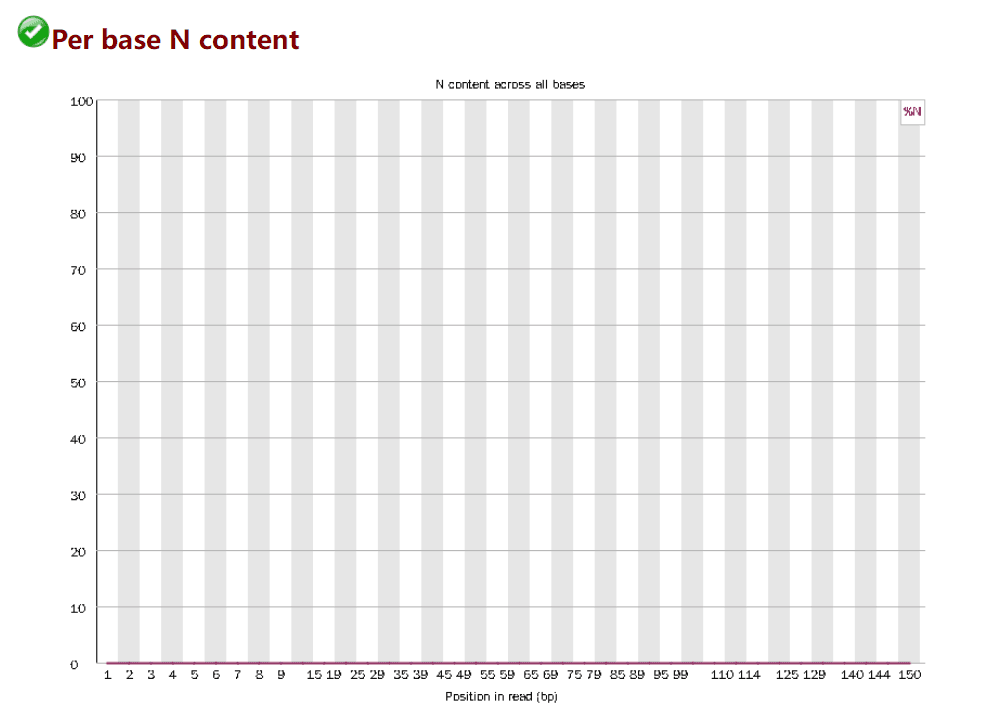
每个位置无法检测的值的比例,当测序仪无法确定是何种碱基时,用 N 表示
- 当任意位置的 N 的比例超过 5%,报"WARN"
- 当任意位置的 N 的比例超过 20%,报"FAIL"
正常情况下,N 的比例是很小的,所以图上常常看到一条直线,但放大 Y 轴之后会发现还是有 N 的存在,这不算问题,当 Y 轴在 0%-100%的范围内也能看到“鼓包”时,说明测序系统出了问题
Sequence Length Distribution
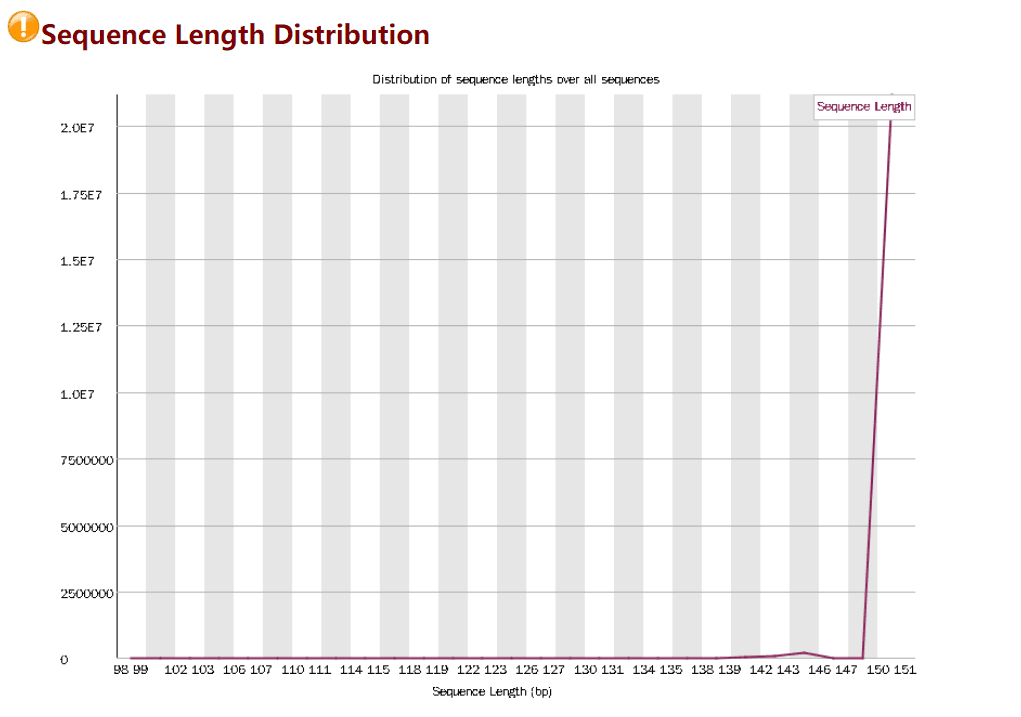
序列测序长度分布
每次测序仪测出来的长度在理论上应该是完全相等的,但是总会有一些偏差,当测序的长度不同时,如果很严重,则表明测序仪在此次测序过程中产生的数据不可信,比如我的这个图中,150pb 是最主要的,其他的几乎没有,所以数据的质量还是比较高的
Sequence Duplication Levels
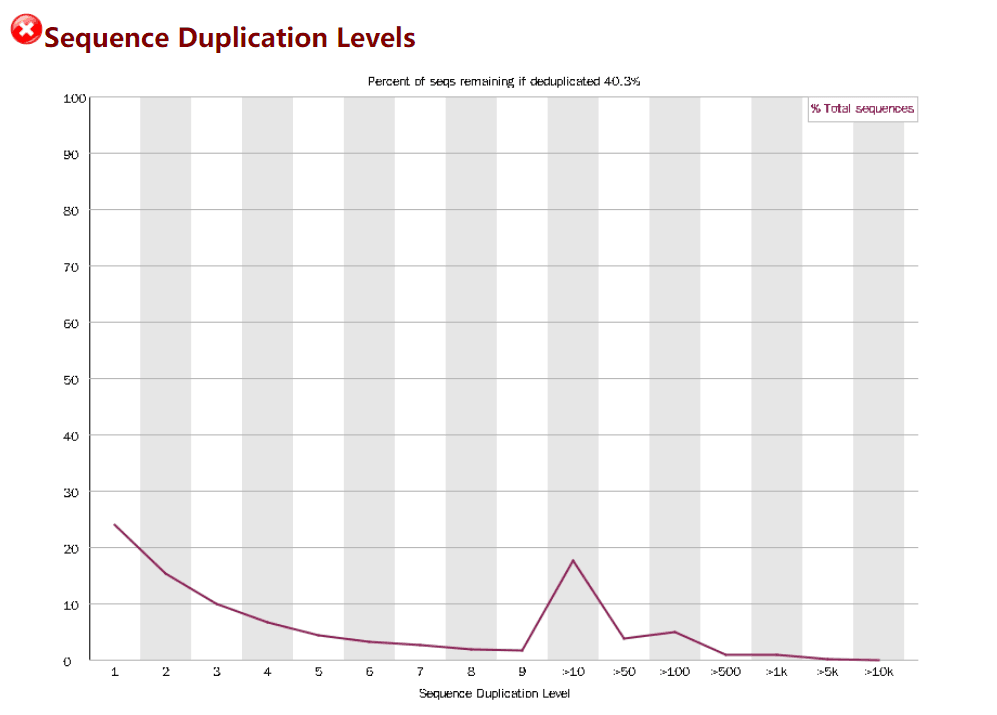
统计 reads 重复水平
- 横坐标是重复的次数
- 纵坐标是 duplicated reads 占 unique reads 总数百分比
- 当非 unique 的 reads 占总数的比例大于 20%时,报"WARN"
- 当非 unique 的 reads 占总数的比例大于 50%时,报"FAIL"
测序本身就会产生重复 reads, 测序深度越高,reads 重复数越大;如果重复出现峰值,就提示可能存在偏差(如建库过程中的 PCR duplication)
fastqc 抽取 reads 文件前 200,000 条 reads 统计其重复情况,重复数目大于等于 10 的 reads 被合并统计,这也是为什么我们看到上图的中间那里略有上扬,大于 75bp 的 reads 只取 50bp 进行比较,由于 reads 越长错误率越高,所以其重复程度仍有可能被低估
Overrepresented sequences

过度重复出现的序列的统计信息,上图中没有
Adapter Content
衡量的是序列中两端 adapter 的情况
如果在当时 fastqc 分析的时候-a 选项没有内容,则默认使用图例中的四种通用 adapter 序列进行统计
上图中 adapter 都已经去除,如果有 adapter 序列没有去除干净的情况,在后续分析的时候需要先使用 cutadapt 等软件进行去接头
Trimmomatic 过滤低质量序列
常用参数
- PE:双端测序文件过滤
- SE:单端测序文件过滤
- -baseout:输出目录
- -threads:线程数,最大是 CPU 核数
- -trimlog:生成日志名
- -quiet:静默模式
- ILLUMINACLIP:从 reads 中剪切 adapter 和其他 Illumina 特定序列
- SLIDINGWINDOW:执行滑动窗口修剪,一旦窗口内的平均质量低于阈值,则切割
- LEADING:如果低于阈值质量,则在 reads 起始处剪切碱基
- TRAILING:如果低于阈值质量,则在 reads 末尾处剪切碱基
- CROP:将 reads 从末尾切割为指定长度
- HEADCROP:从 reads 剪切后低于指定长度,则删除
- MINLEN:如果 reads 低于指定长度,则删除
- TOPHRED33:将质量得分转换为 Phred-33
- TOPHRED64:将质量得分转换为 Phred-64
过滤低质量序列
我使用的是下面的命令,需要根据自己的文件进行调整
trimmomatic PE -threads 1 -phred33 SRR25909836_1.fastq.gz SRR25909836_2.fastq.gz -summary SRR25909836.summary -baseout SRR25909836.fastq.gz LEADING:3 TRAILING:3 SLIDINGWINDOW:5:20 HEADCROP:13 MINLEN:36
解释一下这些参数
- trimmomatic PE: 运行 Trimmomatic 的命令行接口,并指定输入文件是配对的 PE (paired-end) reads
- -threads 1: 指定使用 1 个线程进行处理
- -phred33: 指定输入文件的 Phred 质量值编码是 Phred-33
- SRR25909836_1.fastq.gz 和 SRR25909836_2.fastq.gz: 输入的配对 FASTQ 文件,SRR25909836_1.fastq.gz 是第一条 reads 的文件,SRR25909836_2.fastq.gz 是第二条 reads 的文件
- -summary oryza_sativa.summary: 生成一个名为 oryza_sativa.summary 的文件,其中包含了程序运行的详细统计信息
- -baseout trimmed: 输出文件的基本名称,后面会加上一些后缀来区分不同的输出文件
- LEADING:3: 移除每条 reads 开头的低质量碱基,如果碱基质量低于 3,就被去除
- TRAILING:3: 移除每条 reads 末尾的低质量碱基,如果碱基质量低于 3,就被去除
- SLIDINGWINDOW:5:20: 对每条 reads 进行滑动窗口的质量修剪,如果窗口内的平均质量小于 20,窗口内的所有碱基就会被去除,窗口的大小为 5
- HEADCROP:13: 去除每条 reads 开头的 13 个碱基
- MINLEN:36: 去除所有长度小于 36 的 reads
-phred33 之后的两个是正向和反向的测序文件
会看到五个输出文件 SRR25909836_1P.fastq.gz、SRR25909836_2P.fastq.gz、SRR25909836_1U.fastq.gz、SRR25909836_2U.fastq.gz、oryza_sativa.summary
- SRR25909836_1P.fastq.gz、SRR25909836_2P.fastq.gz 这两个文件包含那些在两个末端都通过质量控制的序列配对,这两个文件是过滤后的测序数据,可以用这两个文件进行后续的测序数据分析
- SRR25909836_1U.fastq.gz、SRR25909836_2U.fastq.gz 两个文件包含那些只有一端通过质量控制的序列,这就是我们说的"单一通过"(unpaired pass)输出
- summary 提供了关于 Trimmomatic 运行的详细信息,它包括以下几类信息
- 输入的读数量
- 裁剪因引物/接头污染或低质量得分而去除的读数量
- 由于长度不足而被丢弃的读物数量
- 输出的读数量
hisat2 比对
hisat2 可以快速准确地将测序得到的 RNA 片段(reads)比对到参考基因组,从而确定这些 RNA 片段在基因组上的精确位置,进一步可以用于基因表达量定量,剪接位点的检测等多种 RNA-Seq 分析任务
建立索引
hisat2 需要一个 index 索引才能进行比对,hisat2 提供了一些 index,但很少,只有人类、小鼠等基因组的,可以在下面的 ftp 地址中进行下载
ftp://ftp.ccb.jhu.edu/pub/infphilo/hisat2/data
由于这里我做的是水稻的,所以就需要自己建立索引,使用的是水稻的参考基因组序列,前面已经下载好了,使用下列命令建立索引
hisat2-build -p 4 oryza_sativa.fa oryza_sativa
- -p 4 是指占用 4 线程
- oryza_sativa.fa 即前面我重命名的参考基因组序列文件
- oryza_sativa 就是参考基因组序列文件,fa 前面的内容
建立完成之后就可以看到系统中多了一个 oryza_sativa 文件夹,cd 进去就可以看到 8 个以 ht2 为拓展名的文件,要将参考基因组序列文件也放到这个文件夹
常用参数
- -x:参考基因组索引文件的前缀
- -1:正向测序文件
- -2:反向测序文件
- -S:输出文件名(sam 文件)
- -P:线程数
- -t:打印加载索引文件和对齐读取所需的时间
- –dta-cufflinks:出来的结果更适合 cufflinks 处理 (主要用于基因表达量的计算和差异表达基因的寻找)
- –no-unal:不记录没比对上的 reads
- –un-conc:在输出文件写出与参考基因组不一致的 reads 对
进行比对
我使用的命令如下(使用经过过滤的测序数据):
hisat2 -x oryza_sativa/oryza_sativa -p 5 -1 SRR25909836_1P.fastq.gz -2 RR25909836_2P.fastq.gz -S SRR25909836.sam
注意-x 后跟索引文件,不加拓展名,保证 ht2 文件和 fa 文件的文件名一致即可,这里由于前面过滤后的序列是没有拓展名的,所以会提示 Warning: Unsupported file format,不影响结果

运行完毕后便得到 sam 文件,还会输出一段信息
z -S oryza_sativa.sam
21675765 reads; of these:
21675765 (100.00%) were paired; of these:
2281601 (10.53%) aligned concordantly 0 times
18927559 (87.32%) aligned concordantly exactly 1 time
466605 (2.15%) aligned concordantly >1 times
----
2281601 pairs aligned concordantly 0 times; of these:
248174 (10.88%) aligned discordantly 1 time
----
2033427 pairs aligned 0 times concordantly or discordantly; of these:
4066854 mates make up the pairs; of these:
2387493 (58.71%) aligned 0 times
1619885 (39.83%) aligned exactly 1 time
59476 (1.46%) aligned >1 times
94.49% overall alignment rate
这些输出记录包含以下信息:
- 总共有 21675765 个读取序列
- 所有读取序列中 100.00%都成对存在(即是成对端 (read pairs) 序列)
- 成对端序列中 10.53%的序列没有成功比对到基因组上
- 87.32%的序列只比对到了基因组上的一个位置
- 2.15%的序列比对到了基因组上的多个位置
- 对于没有成功比对的成对端序列,有 10.88%者的序列不一致地(非正确配对的)比对到了基因组上一个位置
- 有的序列无法一致地或不一致地比对,这些序列占所有没有成功比对的成对端序列的 2033427 对,它们一共包含 4066854 个“pairs”序列
- 在这些“pairs”序列中,
- 58.71%的序列没有比对到任何地方
- 39.83%的序列比对到了基因组上的一个位置
- 1.46%的序列比对到了基因组上的多个位置
- 整体上的比对成功率为 94.49%
在 RNA-Seq 分析中,比对成功率是一个重要的质量控制指标, 94.49%的比对成功率表明,绝大部分读取序列都能够成功地比对到基因组上,这表示 RNA-Seq 实验和测序质量都相对较好
samtools 排序压缩
常用命令
- samtools view:将 SAM 格式文件转换为 BAM 格式,或者执行过滤和查看操作
- -b(输出 BAM 格式)
- -S(输入为 SAM 格式)
- -h(输出头文件)
- samtools sort:对 BAM 文件进行排序
- -o(输出至文件)
- -n(按 read 名称排序)
- samtools index:为排序后的 BAM 文件建立索引
- samtools faidx:为 fasta 格式的参考序列建立索引,并可以快速抽取序列
- samtools tview:文本模式下查看比对结果
- samtools flagstat:提供 BAM 文件的比对统计信息,如总 reads 数,映射的 reads 数等
- samtools mpileup:生成 mpileup 文件用于随后的突变检测
- -u(生成 BCF)
- -g(生成 BCF 或 VCF)
- -f(参考序列)
排序压缩
我使用的是以下命令
samtools sort -n -@ 5 SRR25909836.sam -o SRR25909836.bam
运行完成后会得到一个 bam 文件
featureCounts 生成基因计数表
常用参数
- -a:注释文件的路径,格式为 GTF 或 GFF
- -o:输出文件的路径
- -p:如果序列数据来自于 paired-end RNA-seq,使用此参数
- -B:只保留同时映射到同一基因组特征(例如,基因或外显子)的片段
- -C:不包括由基因间跨越引起的 reads
- -s:设置 strand-specific read 计数,如果使用 0,表示非特异性;如果使用 1,表示第一链特异性;如果使用 2,表示第二链特异性
- -Q:包含比对质量达到特定阈值的 reads,默认值为 10
- -g:制定 GTF 特性类型,例如,gene, transcript 等
- -F:制定输入文件格式,例如,BAM, SAM 等
计数统计
我使用的是以下命令
featureCounts -T 5 -t exon -g Name -a oryza_sativa.gff3 -o counts -p SRR25909836.bam
oryza_sativa.gff3 就是最初下载的注释文件,如果要统计多个文件的话,在-p 后面跟上就可以,会生成 counts、counts.summary 两个文件,
counts.summary 文件是计数统计情况

counts 文件是基因的具体信息

我这里只有一组数据,所以数量统计也只有一列,通常做 RNA-Seq 时是需要多组数据进行分析的,将 bam 文件跟在-p 参数后面即可。