Hi-C—前沿技术带你走进 DNA 的三维空间
本文总阅读量 次
Hi-C 是一种高通量的基因组和表观基因组技术,用于捕获染色质构象。一般来说,Hi-C 被认为是一系列染色体构象捕获技术的衍生物,包括但不限于 3C(染色体构象捕获)、4C(染色体构象捕获-芯片/环状染色体构象捕获)和 5C(染色体构象捕获碳拷贝)。Hi-C 通过结合 3C 和下一代测序(NGS)方法全面检测细胞核中全基因组染色质相互作用,被认为是染色体构象捕获技术(C 技术)发展的质的飞跃,并标志着 3D 基因组学的开端。
基因组是细胞中功能最为关键的部分,它以紧凑的三维(3D)结构进行组织。基因组内含有数百万个核苷酸碱基,这些碱基按照特定的框架排列。随着基因组测序和先进显微镜技术的迅速进步,我们现在能够揭示基因组的 3D 空间结构。利用连接方法的染色体捕获技术以及 3D 基因组浏览器中的可视化工具,研究人员能够更深入地探索基因组。拓扑相关结构域(TADs)、核纤层蛋白相关结构域、CCTCC 结合因子结构域、粘连蛋白以及染色质结构等,都是已被识别并编码基因组 3D 结构的重要成分。

对染色质三维组织在基因表达调控中的作用进行分析的研究正在取得快速进展,这主要归功于技术的突破,如染色体构象捕获技术(3C)、环状染色体构象捕获技术(4C)、碳拷贝染色体构象捕获技术(5C)以及 Hi-C 方法。这些技术已经揭示了基因组的立体组织与细胞核结构之间的相互作用,并表明这种组织可能因细胞类型的不同以及在细胞分化和发育过程中的变化而有所差异。

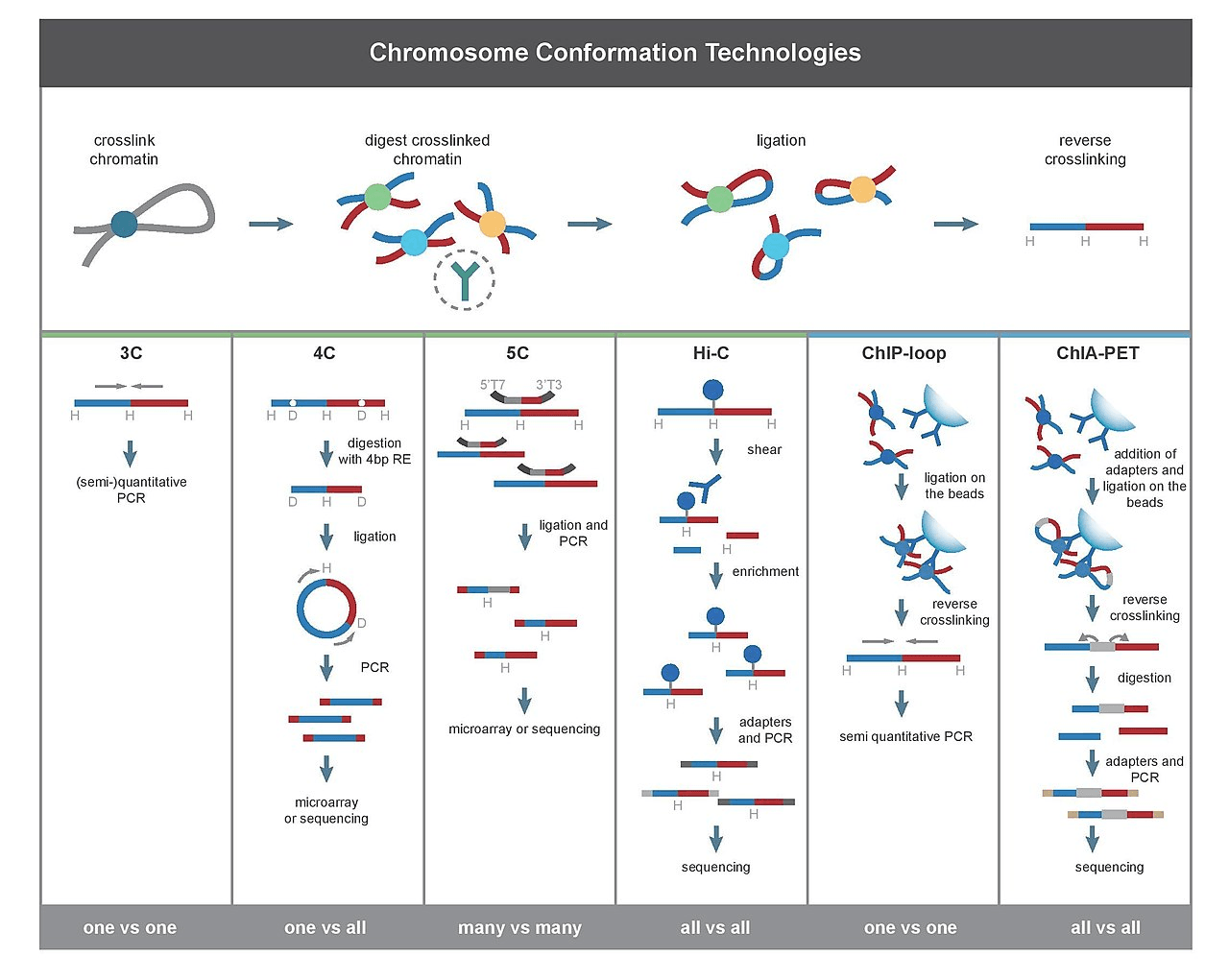
3C(一对一方法)
3C 是一种分析基因组结构的方法,它通过染色质交联和邻近连接的技术来研究不同基因组位点之间的相互作用频率。这种方法需要使用甲醛来固定细胞,确保捕获到由蛋白质和 RNA 介导的接触。固定后,染色质会被限制性内切酶(例如 HindIII 或 DpnII)切割成片段。接着,这些 DNA 片段的相邻末端会被连接起来,并通过 PCR 测序来鉴定那些相互作用的位点。在标准的 3C 实验中,研究者会针对特定的基因座对进行定量 PCR 分析,一次关注一个相互作用对。这样的分析不仅能够揭示出两个基因座之间的直接相互作用,还能够提供关于目标位点的重要先验信息。
4C(一对多方法)
芯片上染色体构象捕获通常称为 4C 或环状染色体构象捕获,对于研究一个区域与基因组其余部分(一个与全部)的相互作用非常有用。环状染色体捕获方法是 3C 技术的一部分,用于解决染色体相互作用的表观遗传控制网络的存在。4C 方法基于邻近连接的原理,其中 DNA-蛋白质/蛋白质-DNA 使用高浓度的连接酶并长时间孵育一周以上,生成环状 DNA 分子。随后,在连接过程中反转靠近靶序列的交联引物,以物理接近的方式扩增 DNA,而无需事先了解其身份。该过程能够扩增交联染色质中各种大小的序列。4C 采用与 3C 相同的技术来获得连接产物。限制性产物与 3C 模板连接并孵育过夜,同时频繁切割第二种限制性酶。随后,生成线性序列作为 4C 模板进行引物杂交。根据标准免疫沉淀(ChIP)方案将这些 4C 模板与阵列杂交。通过 4C 原理,可以揭示活性和非活性染色质结构域的核组织。此外,可以使用 4C 分析来研究 SOX9 启动子的长程顺式相互作用。所有触点都可以使用 4C 原理映射到单个位点。可以使用某个区域的引物(启动子/增强子)来扩增该位点的所有连接伙伴,然后对扩增产物进行测序(每个文库的深度为 1-5 百万个读数)。这有助于以几千碱基的分辨率分析感兴趣区域的全基因组相互作用伙伴。该程序非常适合检测短程调控相互作用、长基因组距离和整个染色体的相互作用。
5C(多种方法与全部方法)
5C 技术可以用来绘制大型基因组区域内的所有接触点的图谱。在 5C 中,使用正向和反向引物扩增 3C 文库中高达数兆碱基的大基因组区域。5C 可以扩增大型基因组区域,以获得 1 Mb 区域的 15-20 kb 分辨率。然而,5C 的分辨率取决于对特定位点的正向和反向引物的设计。如果没有适当的引物,就必须从接触图谱中排除可映射的片段。5C 技术可用于研究特定目标位点的复杂相互作用,但不适用于研究全基因组相互作用,因为它需要数百万个 5C 引物。循环相互作用可以使用 5C 方法进行研究,该方法可广泛应用于绘制基因组元件顺反相互作用网络和研究高阶染色体结构。
Hi-C(全部与全部方法)
Hi-C 测量两个 DNA 片段在 3D 空间中物理关联的频率(作为细胞群体的平均值),将染色体结构直接连接到基因组序列。 Hi-C 的一般程序包括首先使用甲醛交联染色质材料。然后,染色质被溶解并片段化,相互作用的基因座被重新连接在一起以产生嵌合 DNA 分子的基因组文库。 这些嵌合体或连接产物的相对丰度与各个染色质片段在细胞群中在 3D 空间中相互作用的概率相关。 3C 专注于分析一组预定的基因组位点,以提供对感兴趣的染色体区域构象的“一对一些”研究,Hi-C 通过在连接前用生物素化的核苷酸标记所有片段化的染色质,实现“全对全”相互作用谱分析。 因此,生物素标记的连接点可以通过链霉亲和素包被的磁珠更有效地纯化,并且染色质相互作用数据可以通过 Hi-C 文库的直接测序获得。
ChIP-loop
该技术将 3C 与 ChIP-seq 结合,可以检测目的蛋白质介导的两个目的基因区域互作。
ChIA-PET
该技术将 HiC 与 ChIP-seq 结合,可以检测目的蛋白质的所有互相作用。
| 互作 | 覆盖 | 作用 | |
|---|---|---|---|
| 3C | 单对单 | <1Mb | 检测已知基因组基因区域之间的互相作用 |
| 4C | 一对多 | 全基因组 | 确定某基因组区域与其他区域的互相作用 |
| 5C | 多对多 | <1Mb | 确定染色体特定区域内的全部高级结构 |
| Hi-C | 全部互作 | 全基因组 | 检测全基因组范围内的全部高级结构 |
| ChIP-loop | 一对一 | <1Mb | 检测目的蛋白质介导的两个目的基因区域互作 |
| ChIA-PET | 全部互作 | 全基因组 | 检测目的蛋白质的所有互相作用 |
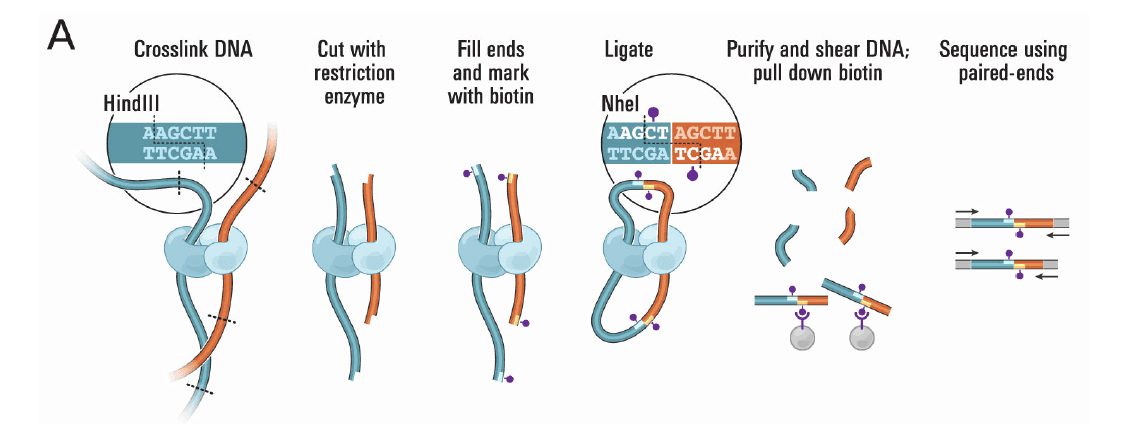
Hi-C 技术流程
- 通过甲醛交联固定,将细胞内由蛋白质介导的空间上邻近的染色质片段进行共价连接。
- 限制性内切酶进行酶切
- 使用生物素标记末端标记
- 将连接的 DNA 纯化后超声打断,并用生物素亲和层析,将生物素化的 DNA 片段分离,加上接头进行高通量测序
Hi-C 技术的应用
- 量化在三维空间中基因组的染色质间交联(cross-linked chromatin )
- 解析全基因组互作模式,如启动子和增强子互作
- 构建三维空间结构模型,如研究基因组三维结构特征:compartment,TAD,loop 等
- 构建全基因组互作图谱
- 辅助提升基因组组装
- 构建基因组单体型图谱