点点点!TBtools 完成论文复现
本文总阅读量 次
文章题为"Identification and expression pattern analysis of the OsSnRK2 gene family in rice"。该文章发表于 Frontiers in Plant Science(中科院二区,IF=5.6,第一作者:Tongyuan Yu,通讯作者:Dawei Xue)。
根据原文的材料与方法,文章做了以下生信分析:
- domain 分析
- 染色体定位
- 基因结构分析
- motif 分析
- 系统发育树
- 共线性分析
- 多序列比对
- 蛋白质理化性质分析
- 蛋白质二、三级结构预测
- 互作蛋白网络
- 顺式作用元件分析
- 表达热图
以上的分析基本都可以用 TBtools 来完成并做成好看的图片,这也是一个常规基因家族分析的流程。此处不涉及过于详细的步骤,更详细的步骤请参阅我的博客文章:https://yuanj.top/posts/a4559e49
作者在补充材料里提供了基因 ID、基因名和蛋白序列:
>OsSAPK1
MERYEVMRDIGSGNFGVAKLVRDVATNHLFAVKFIERGLKIDEHVQREIMNHRSLKHPNIIRFKEVVLTPTHLAIVMEYAAGGELFERICNAGRFSEDEARFFFQQLISGVSYCHSMQVCHRDLKLENTLLDGSVTPRLKICDFGYSKSSVLHSQPKSTVGTPAYIAPEVLSRKEYDGKVADVWSCGVTLYVMLVGAYPFEDPDDPRNFRKTITRILSVQYSIPDYVRVSADCRHLLSRIFVGNPEQRITIPEIKNHPWFLKNLPIEMTDEYQRSMQLADMNTPSQSLEEVMAIIQEARKPGDAMKLAGAGQVACLGSMDLDDIDDIDDIDIENSGDFVCAL
>OsSAPK2
MERYEVIKDIGSGNFGVAKLVRDVRTKELFAVKFIERGQKIDENVQREIMNHRSLRHPNIVRFKEVVLTPTHLAIVMEYAAGGELFERICSAGRFSEDEARFFFQQLISGVSYCHSMQICHRDLKLENTLLDGSIAPRLKICDFGYSKSSLLHSQPKSTVGTPAYIAPEVLARKEYDGKVADVWSCGVTLYVMLVGAYPFEDPDEPRNFRKTITRILSVQYMVPDYVRVSMECRHLLSRIFVANPEQRITIPEIKNHPWFLKNLPIEMTDEYQMSVQMNDINTPSQGLEEIMAIIQEARKPGDGSKFSGQIPGLGSMELDDVDTDDIDVEDSGDFVCAL
>OsSAPK3
MEERYEALKELGAGNFGVARLVRDKRSKELVAVKYIERGKKIDENVQREIINHRSLRHPNIIRFKEVCLTPTHLAIVMEYAAGGELFEQICTAGRFSEDEARYFFQQLISGVSYCHSLEICHRDLKLENTLLDGSPTPRVKICDFGYSKSALLHSKPKSTVGTPAYIAPEVLSRKEYDGKATDVWSCGVTLYVMLVGSYPFEDPGDPRNFRKTISRILGVQYSIPDYVRVSSDCRRLLSQIFVADPSKRITIPEIKKHTWFLKNLPKEISEREKADYKDTDAAPPTQAVEEIMRIIQEAKVPGDMAAADPALLAELAELKSDDEEEAADEYDTY
>OsSAPK4
MEKYEAVRDIGSGNFGVARLMRNRETRELVAVKCIERGHRIDENVYREIINHRSLRHPNIIRFKEVILTPTHLMIVMEFAAGGELFDRICDRGRFSEDEARYFFQQLICGVSYCHHMQICHRDLKLENVLLDGSPAPRLKICDFGYSKSSVLHSRPKSAVGTPAYIAPEVLSRREYDGKLADVWSCGVTLYVMLVGAYPFEDQDDPKNIRKTIQRIMSVQYKIPDYVHISAECKQLIARIFVNNPLRRITMKEIKSHPWFLKNLPRELTETAQAMYYRRDNSVPSFSDQTSEEIMKIVQEARTMPKSSRTGYWSDAGSDEEEKEEEERPEENEEEEEDEYDKRVKEVHASGELRMSSLRI
>OsSAPK5
MEKYEPVREIGAGNFGVAKLMRNKETRELVAMKFIERGNRIDENVFREIVNHRSLRHPNIIRFKEVVVTGRHLAIVMEYAAGGELFERICEAGRFHEDEARYFFQQLVCGVSYCHAMQICHRDLKLENTLLDGSPAPRLKICDFGYSKSSLLHSRPKSTVGTPAYIAPEVLSRREYDGKLADVWSCGVTLYVMLVGAYPFEDPKDPKNFRKTISRIMSVQYKIPEYVHVSQPCRHLLSRIFVANPYKRISMGEIKSHPWFLKNLPRELKEEAQAVYYNRRGADHAASSASSAAAAAAFSPQSVEDIMRIVQEAQTVPKPDKPVSGYGWGTDDDDDDQQPAEEEDEEDDYDRTVREVHASVDLDMSNLQIS
>OsSAPK6
MEKYELLKDIGSGNFGVARLMRNRETKELVAMKYIPRGLKIDENVAREIINHRSLRHPNIIRFKEVVLTPTHLAIVMEYAAGGELFDRICSAGRFSEDESRYFFQQLICGVSYCHFMQICHRDLKLENTLLDGSPAPRLKICDFGYSKSSLLHSKPKSTVGTPAYIAPEVLSRREYDGKMADVWSCGVTLYVMLVGAYPFEDPDDPKNFRKTIGRIVSIQYKIPEYVHISQDCRQLLSRIFVANPAKRITIREIRNHPWFMKNLPRELTEAAQAKYYKKDNSARTFSDQTVDEIMKIVQEAKTPPPSSTPVAGFGWTEEEEQEDGKNPDDDEGDRDEEEGEEGDSEDEYTKQVKQAHASCDLQKS
>OsSAPK7
MERYELLKDIGAGNFGVARLMRNKETKELVAMKYIPRGLKIDENVAREIINHRSLRHPNIIRFKEVVVTPTHLAIVMEYAAGGELFDRICNAGRFSEDEARYFFQQLICGVSYCHFMQICHRDLKLENTLLDGSPAPRLKICDFGYSKSSLLHSKPKSTVGTPAYIAPEVLSRREYDGKTADVWSCGVTLYVMLVGAYPFEDPDDPKNFRKTIGRIMSIQYKIPEYVHVSQDCRQLLSRIFVANPAKRITIREIRNHPWFLKNLPRELTEAAQAMYYKKDNSAPTYSVQSVEEIMKIVEEARTPPRSSTPVAGFGWQEEDEQEDNSKKPEEEQEEEEDAEDEYDKQVKQVHASGEFQLS
>OsSAPK8
MAAAGAGAGAPDRAALTVGPGMDMPIMHDSDRYELVRDIGSGNFGVARLMRDRRTMELVAVKYIERGEKIDDNVQREIINHRSLKHPNIIRFKEVILTPTHLAIVMEYASGGELFERICKNVRFSEDEARYFFQQLISGVSYCHSMQVCHRDLKLENTLLDGSPAPRLKICDFGYSKSSVLHSQPKSTVGTPAYIAPEVLLKKEYDGKTADVWSCGVTLYVMVVGAYPFEDPEEPKNFRKTIQRILNVQYSIPENVDISPECRHLISRIFVGDPSLRITIPEIRSHGWFLKNLPADLMDDDSMSSQYEEPDQPMQTMDQIMQILTEATIPPACSRINHILTDGLDLDDDMDDLDSDSDIDVDSSGEIVYAM
>OsSAPK9
MERAAAGPLGMEMPIMHDGDRYELVKEIGSGNFGVARLMRNRASGDLVAVKYIDRGEKIDENVQREIINHRSLRHPNIIRFKEVILTPTHLAIVMEYASGGELFERICSAGRFSEDEARFFFQQLISGVSYCHSMQVCHRDLKLENTLLDGSTAPRLKICDFGYSKSSVLHSQPKSTVGTPAYIAPEVLLKKEYDGKIADVWSCGVTLYVMLVGAYPFEDPEDPKNFRKTIQKILGVQYSIPDYVHISPECRDLITRIFVGNPASRITMPEIKNHPWFMKNIPADLMDDGMVSNQYEEPDQPMQNMNEIMQILAEATIPAAGTSGINQFLTDSLDLDDDMEDMDSDLDLDIESSGEIVYAM
>OsSAPK10
MDRAALTVGPGMDMPIMHDGDRYELVRDIGSGNFGVARLMRSRADGQLVAVKYIERGDKIDENVQREIINHRSLRHPNIIRFKEVILTPTHLAIVMEYASGGELFERICNAGRFSEDEARFFFQQLISGVSYCHSMQVCHRDLKLENTLLDGSTAPRLKICDFGYSKSSVLHSQPKSTVGTPAYIAPEVLLKKEYDGKIADVWSCGVTLYVMLVGAYPFEDPDEPKNFRKTIQRILGVQYSIPDYVHISPECRDLIARIFVANPATRISIPEIRNHPWFLKNLPADLMDDSKMSSQYEEPEQPMQSMDEIMQILAEATIPAAGSGGINQFLNDGLDLDDDMEDLDSDPDLDVESSGEIVYAM
domain 分析
结构域(domain)是位于超二级结构和三级结构间的一个层次。结构域是在蛋白质的三级结构内的独立折叠单元,通常都是几个超二级结构单元的组合。在较大的蛋白质分子中,由于多肽链上相邻的超二级结构紧密联系,进一步折叠形成一个或多个相对独立的致密三维实体,即结构域。
结构域实质上是二级结构的组合体,充当三级结构的构件。每个结构域分别代表一种功能。
原文中基因家族是指水稻中一系列具有 S_TKc 结构域的基因,是磷酸转移酶,丝氨酸或苏氨酸特异性激酶亚家族。我们可以在 SMART 网站查看该结构域的信息:S_TKc。补充材料里面也提供了基因的蛋白序列,我们直接拿过来用。将蛋白序列粘贴到 SMART 网站 查看结构域:
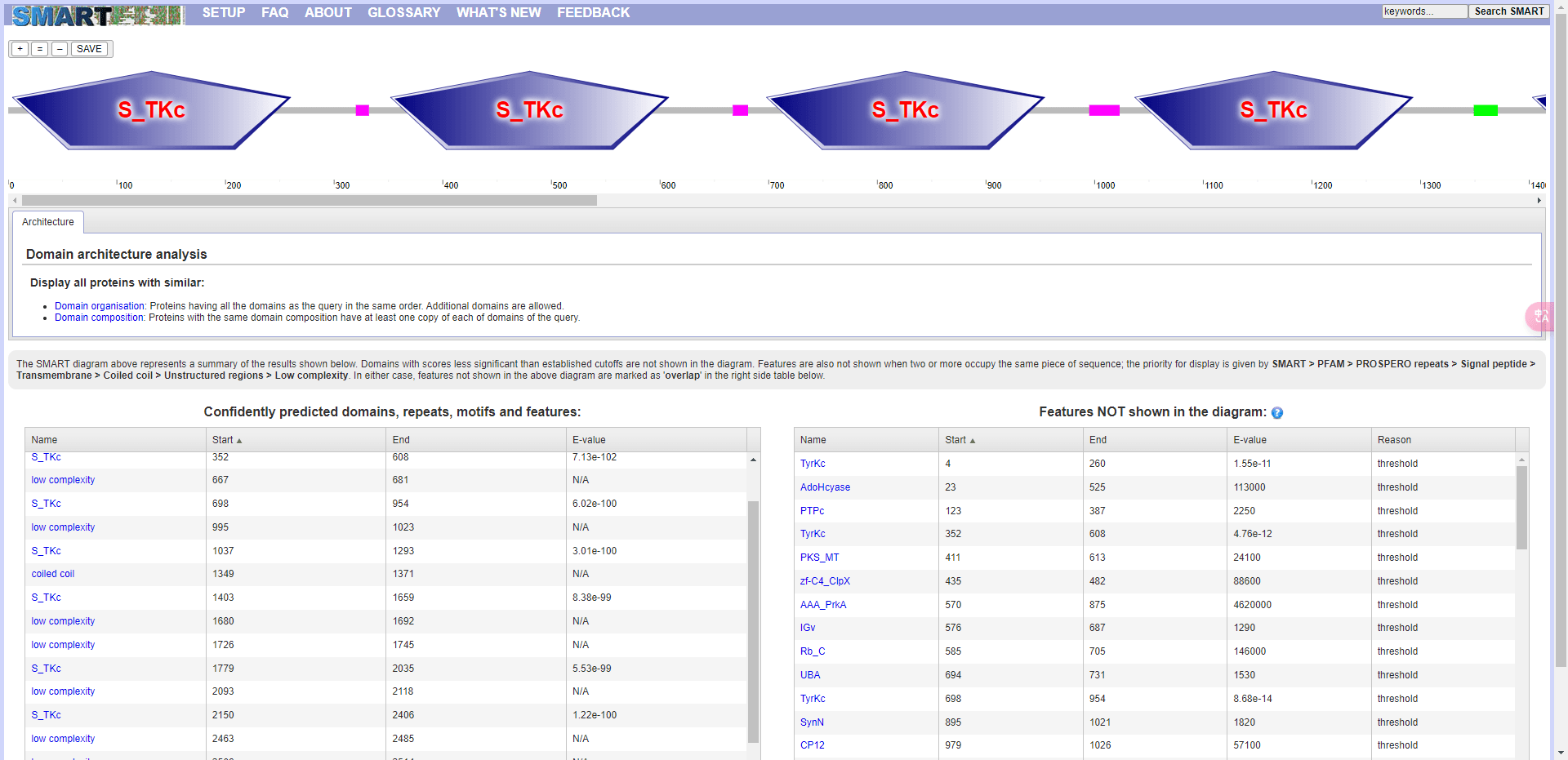
结果显示所有的基因都是具有 S_TKc 结构域的。但是个人感觉 SMART 网站的结果可能看上去不是很友好,我喜欢用 NCBI CD-search,也是上传序列开始分析即可,显示效果更加直观:
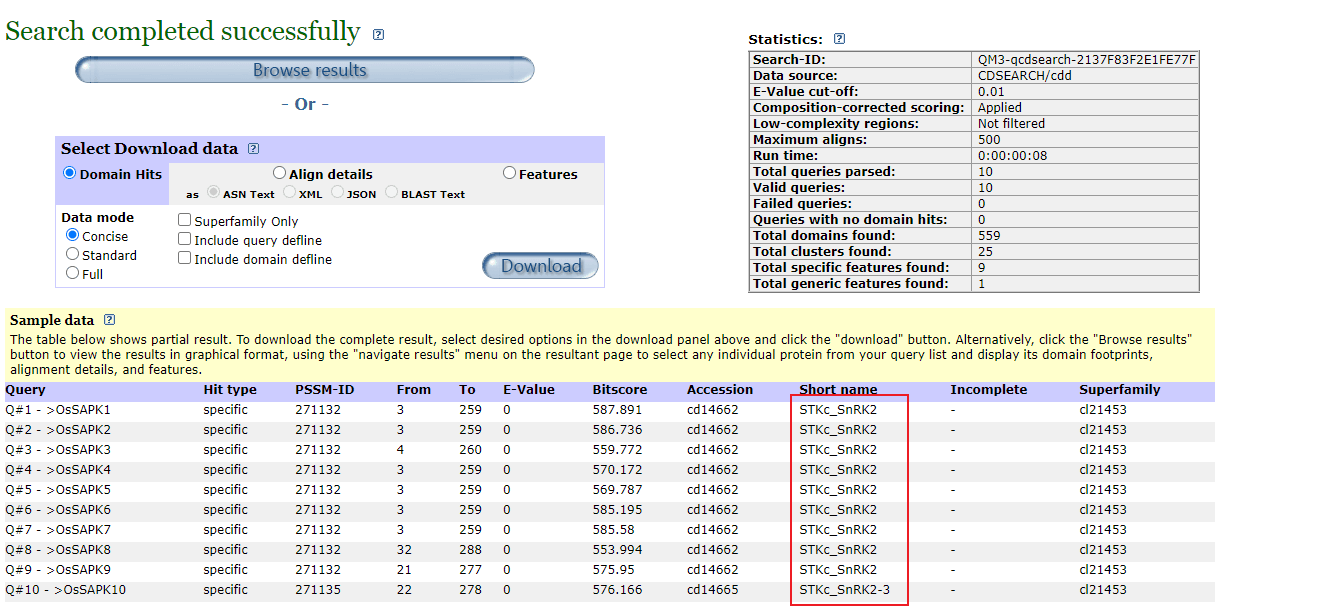
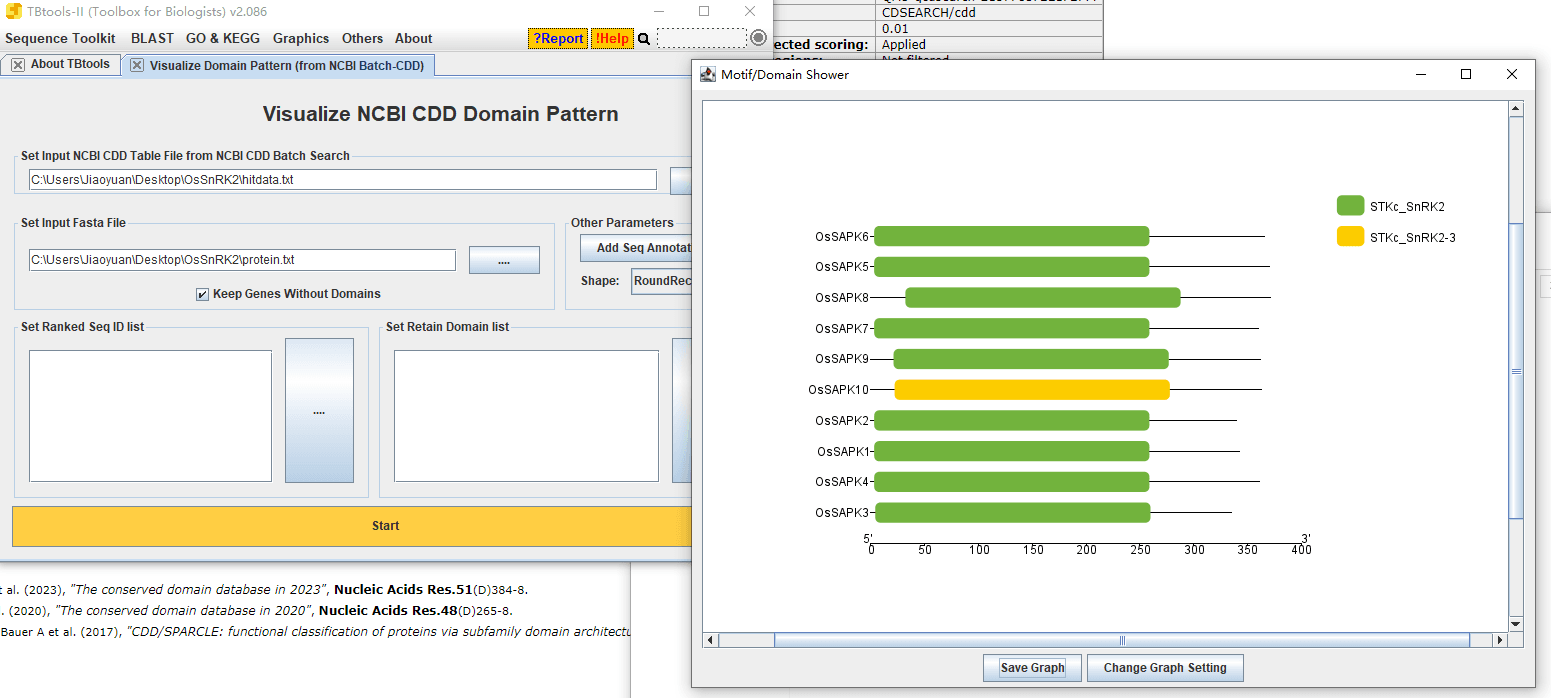
并且也可以用 TBtools 做成图:
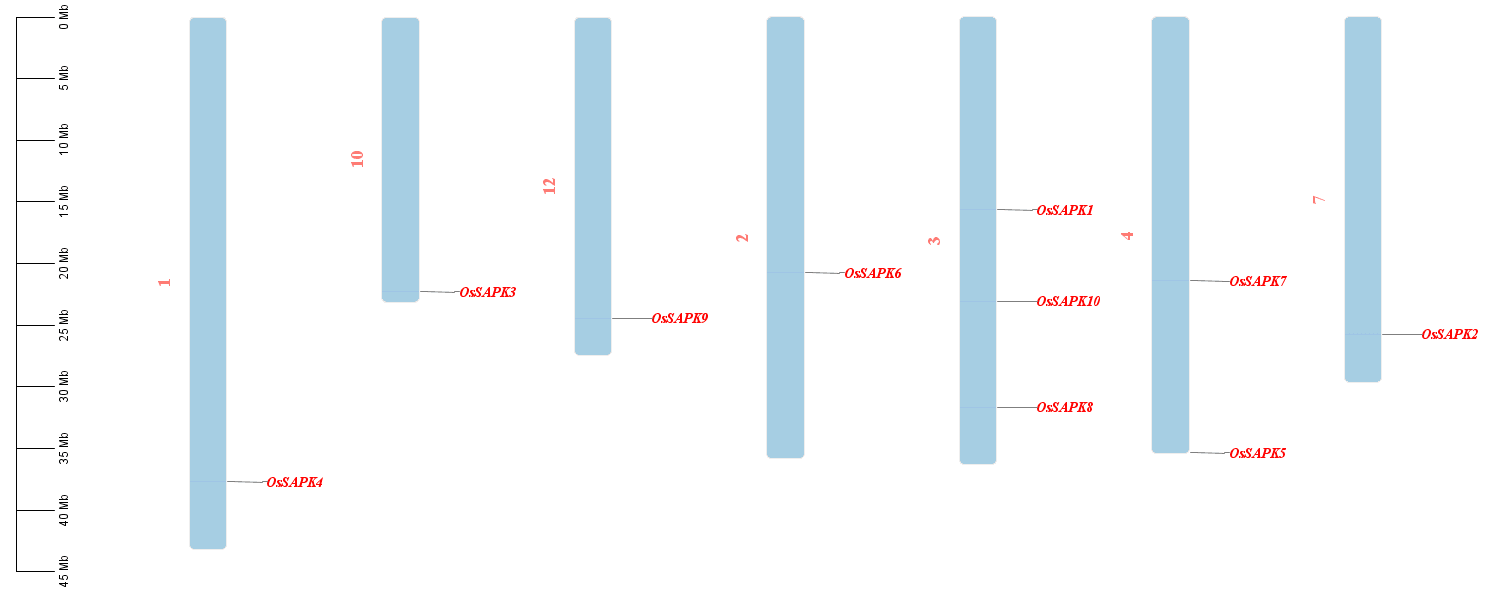
染色体定位
从 Ensembl plants 下载水稻(日本晴)基因组的注释文件,然后将基因 ID 写在一个文件中:
transcript:Os03t0390200-01
transcript:Os07t0622000-01
transcript:Os10t0564500-01
transcript:Os01t0869900-01
transcript:Os04t0691100-01
transcript:Os02t0551100-01
transcript:Os04t0432000-01
transcript:Os03t0764800-01
transcript:Os12t0586100-01
transcript:Os03t0610900-01
再准备一个 rename 文件:
transcript:Os03t0390200-01 OsSAPK1
transcript:Os07t0622000-01 OsSAPK2
transcript:Os10t0564500-01 OsSAPK3
transcript:Os01t0869900-01 OsSAPK4
transcript:Os04t0691100-01 OsSAPK5
transcript:Os02t0551100-01 OsSAPK6
transcript:Os04t0432000-01 OsSAPK7
transcript:Os03t0764800-01 OsSAPK8
transcript:Os12t0586100-01 OsSAPK9
transcript:Os03t0610900-01 OsSAPK10
这里因为 Ensembl plants 下载的注释文件中默认的基因 ID 是转录本的,所以需要对 ID 进行一些处理,加上transcript:再把 Os 的 ID 转换成转录本 ID 即可。
导入到 TBtools:
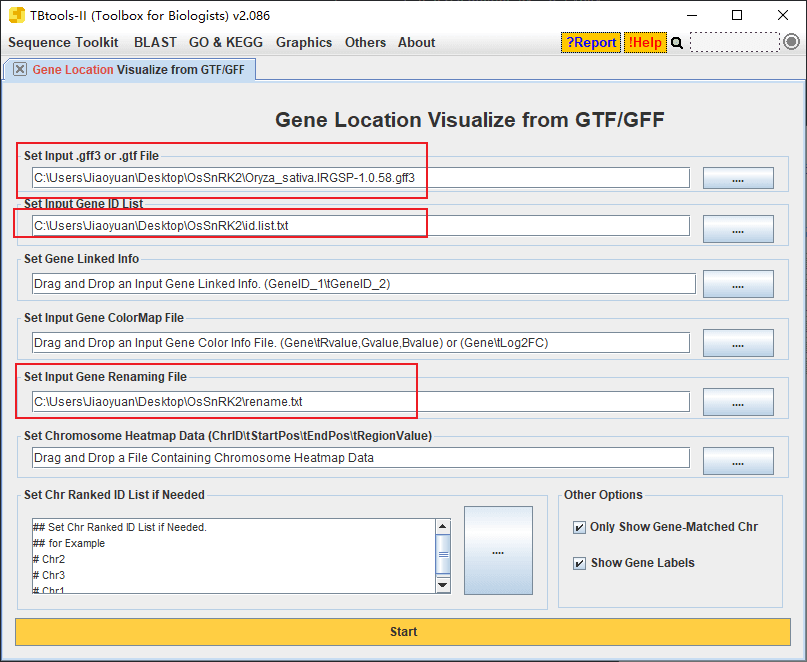
出图后美化一下:
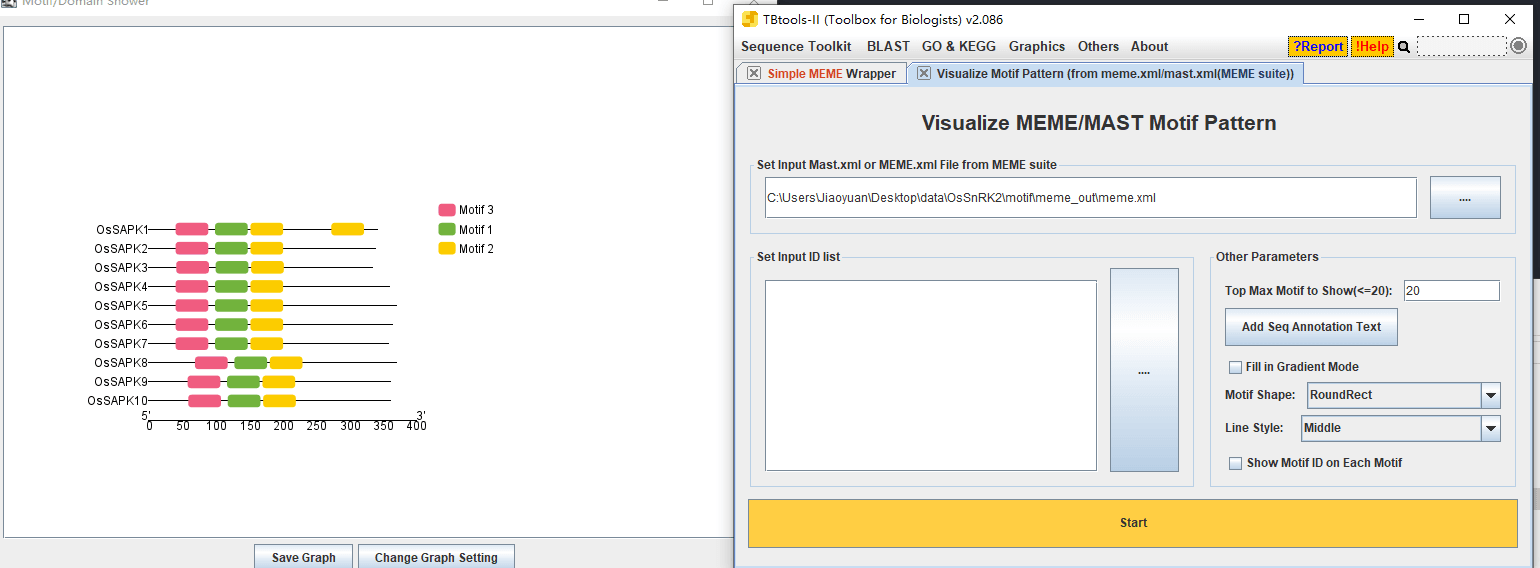
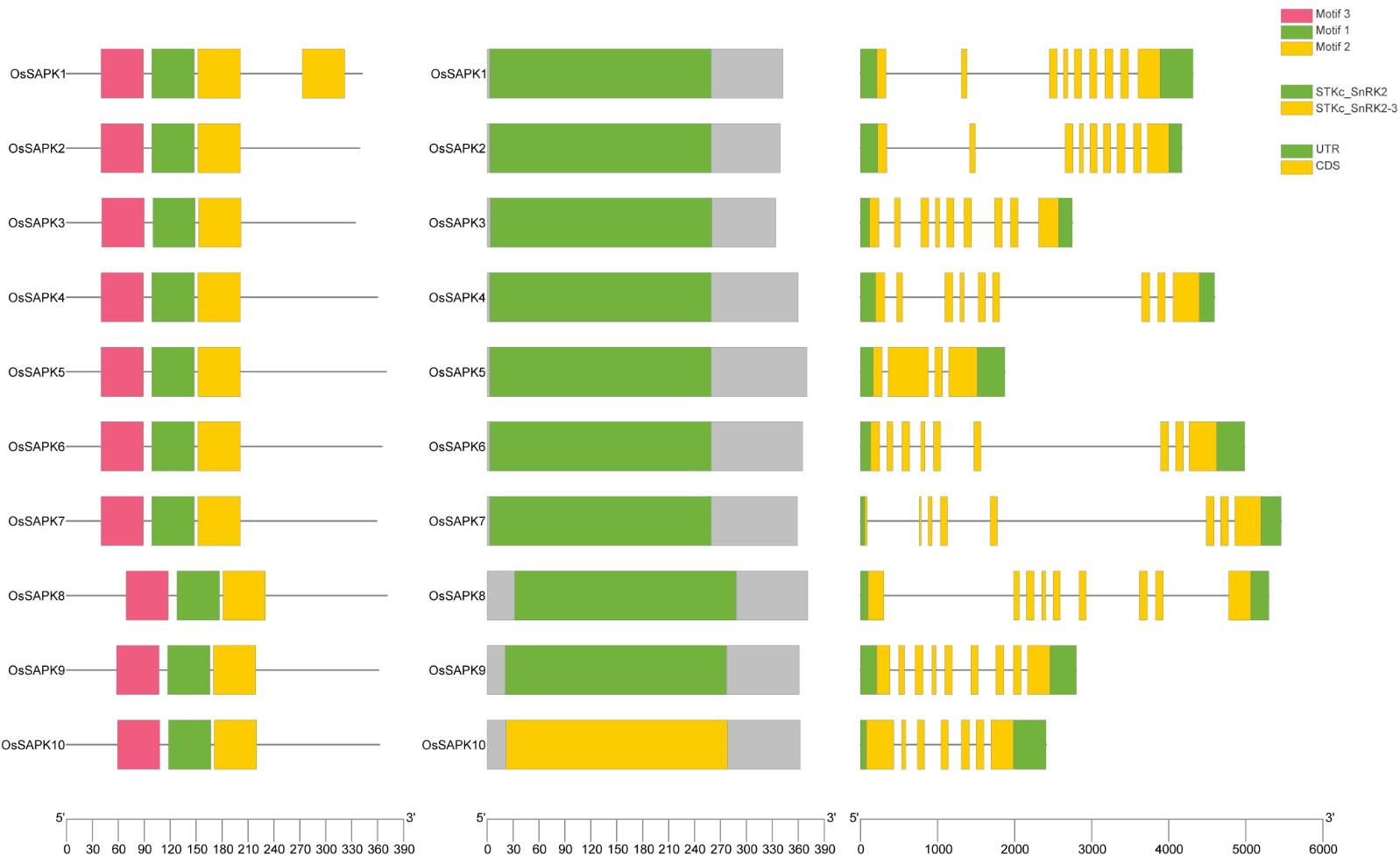
基因结构分析&motif 分析
将蛋白序列上传到 meme 网站 后下载输出的 xml 文件即可,但是网页版 MEME Suite 经常要排队,所以可以用 TBtools 进行本地分析:
再将输出的 xml 文件可视化:
通常我们将基因结构、domain、motif 展示在一张图上:
系统发育树
作者将拟南芥和大麦中的 SnRK2 家族基因也放在一起进行了系统发育分析,我在文章"# Genome-wide identification and expression pattern of SnRK gene family under several hormone treatments and its role in floral scent emission in Hedychium coronarium“中找到了 10 个拟南芥 SnRK2 基因,在文章"Genome-Wide Identification, Expression Pattern and Sequence Variation Analysis of SnRK Family Genes in Barley"中找到 11 个大麦 SnRK2 基因,提取其蛋白质序列共同进行系统发育分析:
| Gene Name | Gene ID |
|---|---|
| AtSnRK1.1 | At3g01090 |
| AtSnRK1.2 | At3g29160 |
| AtSnRK1.3 | At5g39440 |
| AtSnRK2.1 | At5g08590 |
| AtSnRK2.2 | At3g50500 |
| AtSnRK2.3 | At5g66880 |
| AtSnRK2.4 | At1g10940 |
| AtSnRK2.5 | At5g63650 |
| AtSnRK2.6 | At4g33950 |
| AtSnRK2.7 | At4g40010 |
| AtSnRK2.8 | At1g78290 |
| AtSnRK2.9 | At2g23030 |
| AtSnRK2.10 | At1g60940 |
| Gene Name | Gene ID |
|---|---|
| HvSnRK1.1 | HORVU1Hr1G081310 |
| HvSnRK1.2 | HORVU3Hr1G069190 |
| HvSnRK1.3 | HORVU3Hr1G107990 |
| HvSnRK1.4 | HORVU4Hr1G056610 |
| HvSnRK2.1 | HORVU1Hr1G055340 |
| HvSnRK2.2 | HORVU1Hr1G074670 |
| HvSnRK2.3 | HORVU2Hr1G029900 |
| HvSnRK2.4 | HORVU2Hr1G075470 |
| HvSnRK2.5 | HORVU2Hr1G110230 |
| HvSnRK2.6 | HORVU2Hr1G125950 |
| HvSnRK2.7 | HORVU3Hr1G082690 |
| HvSnRK2.8 | HORVU4Hr1G013540 |
| HvSnRK2.9 | HORVU5Hr1G018340 |
| HvSnRK2.10 | HORVU5Hr1G097630 |
| HvSnRK2.11 | HORVU0Hr1G011570 |
使用 Mega7 分析完之后使用 iTOL 网站 进行美化:
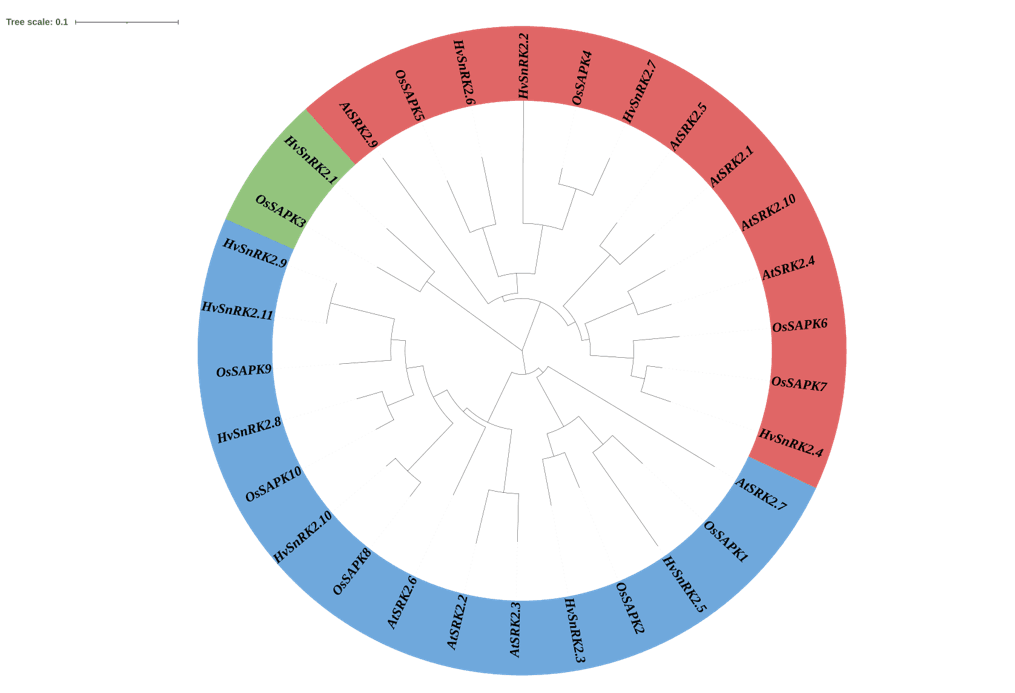
关于进化树的分析步骤及美化可以参阅:https://yuanj.top/posts/8c3c2e59/
共线性分析
共线性分析的步骤比较多,这里不展开赘述,参考我的博客文章:https://yuanj.top/posts/f44e9fb8/
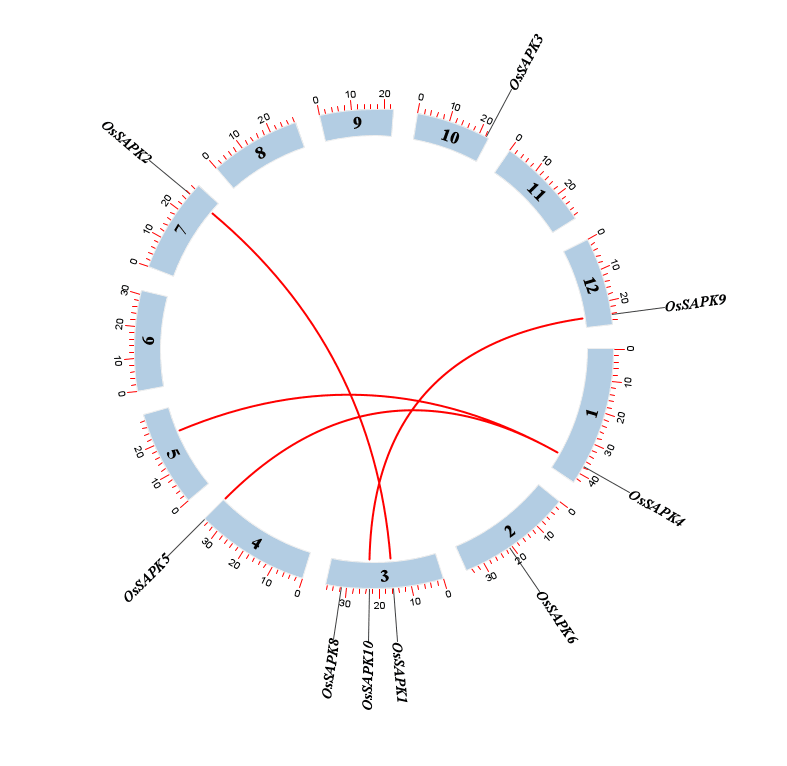
多序列比对
多序列比对我一般用 CLUSTALW 和 ESPript 来做,将蛋白序列上传到 CLUSTALW 进行多序列比对,下载 aln 的结果后用 ESPript 进行可视化:

蛋白质理化性质分析
用 TBtools 即可一键分析,导入蛋白质序列即可:
| Sequence ID | Number of Amino Acid | Molecular Weight | Theoretical pI | Instability Index | Aliphatic Index | Grand Average of Hydropathicity |
|---|---|---|---|---|---|---|
| OsSAPK1 | 342 | 38698.3 | 5.43 | 27.49 | 89.47 | -0.208 |
| OsSAPK2 | 339 | 38538.14 | 5.31 | 32.18 | 87.94 | -0.248 |
| OsSAPK3 | 334 | 37873.18 | 5.67 | 37.29 | 85.87 | -0.424 |
| OsSAPK4 | 360 | 41978.75 | 6.06 | 53.65 | 77.97 | -0.616 |
| OsSAPK5 | 370 | 42165.77 | 5.99 | 42.18 | 77.22 | -0.501 |
| OsSAPK6 | 365 | 41803.36 | 5.72 | 43.13 | 74.27 | -0.621 |
| OsSAPK7 | 359 | 41324.97 | 5.83 | 45.89 | 77.41 | -0.567 |
| OsSAPK8 | 371 | 41723.44 | 4.85 | 44.33 | 86.71 | -0.303 |
| OsSAPK9 | 361 | 40628.32 | 4.81 | 35.85 | 85.62 | -0.272 |
| OsSAPK10 | 362 | 40699.27 | 4.8 | 40.36 | 87.54 | -0.281 |
蛋白质二、三级结构预测
原文作者使用 SOPMA 在线网站 进行蛋白质二级结构预测,直接提交序列就可以得到结果:
SOPMA :
Alpha helix (Hh) : 122 is 35.99%
310 helix (Gg) : 0 is 0.00%
Pi helix (Ii) : 0 is 0.00%
Beta bridge (Bb) : 0 is 0.00%
Extended strand (Ee) : 62 is 18.29%
Beta turn (Tt) : 22 is 6.49%
Bend region (Ss) : 0 is 0.00%
Random coil (Cc) : 133 is 39.23%
Ambiguous states (?) : 0 is 0.00%
Other states : 0 is 0.00%
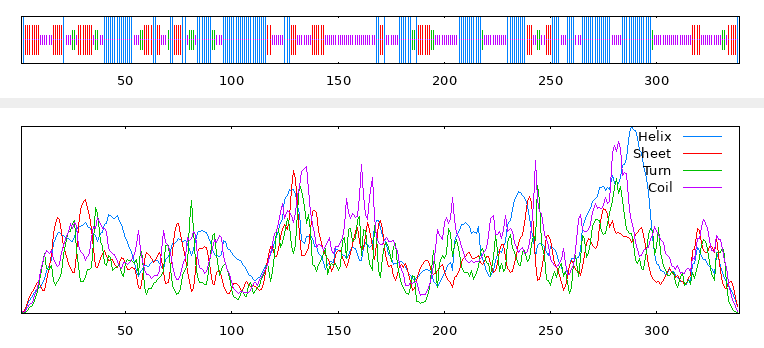
我这里仅仅分析了 OsSAPK2 的二级结构。
三级结构用 UniProt 网站,直接搜索蛋白的 ID 即可:
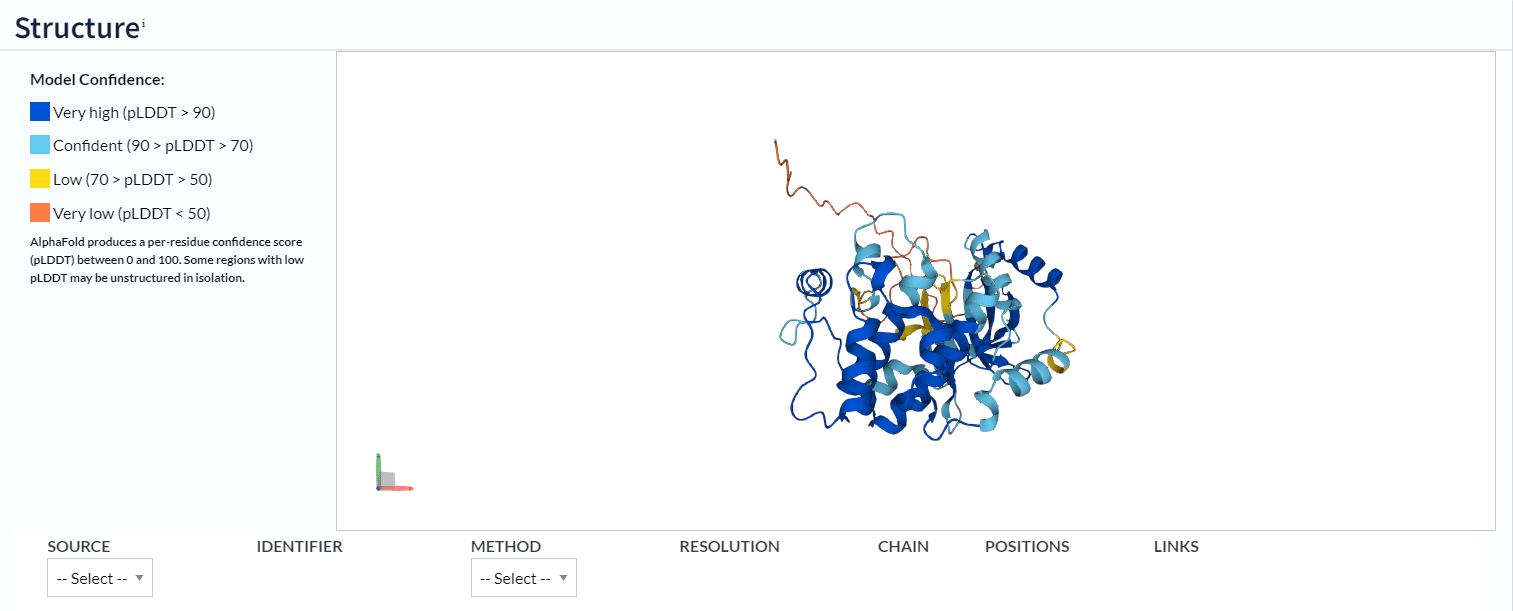
这里我也同样只分析了 OsSAPK2 的三级结构。
互作蛋白网络
互作蛋白网络使用 STRING 数据库完成,直接输入基因 ID 和物种即可:
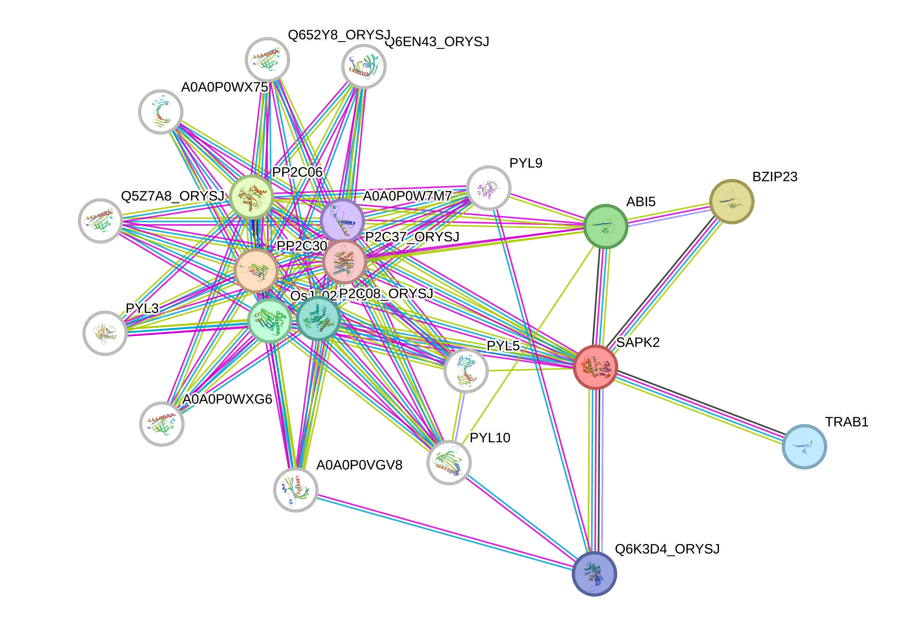
将数据导出为 tsv 文件后,可以使用 Cytoscape 进行美化,可以参照我的博客文章:https://yuanj.top/posts/2eef5da0/ 之前 STRING 数据库默认的互作蛋白网络不够美观,但是现在已经更新,默认的图已经很漂亮了,此处就不进行美化了。
顺式作用元件分析
将上游 2000 的 cds 序列上传到 plantcare 网站,拿到结果后处理一下数据然后导入 TBtools 就可以进行绘图,详细步骤可以参考我的博客文章:https://yuanj.top/posts/6c282499/
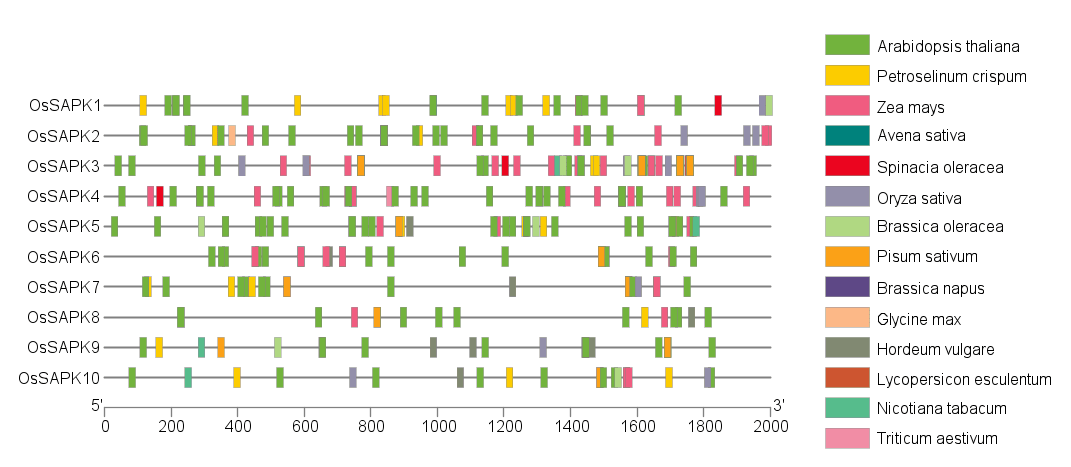
表达热图
原文的表达谱数据是从 CREP 数据库 下载的,同样从 CREP 数据库下载表达量数据后用 TBtools 绘制热图即可:
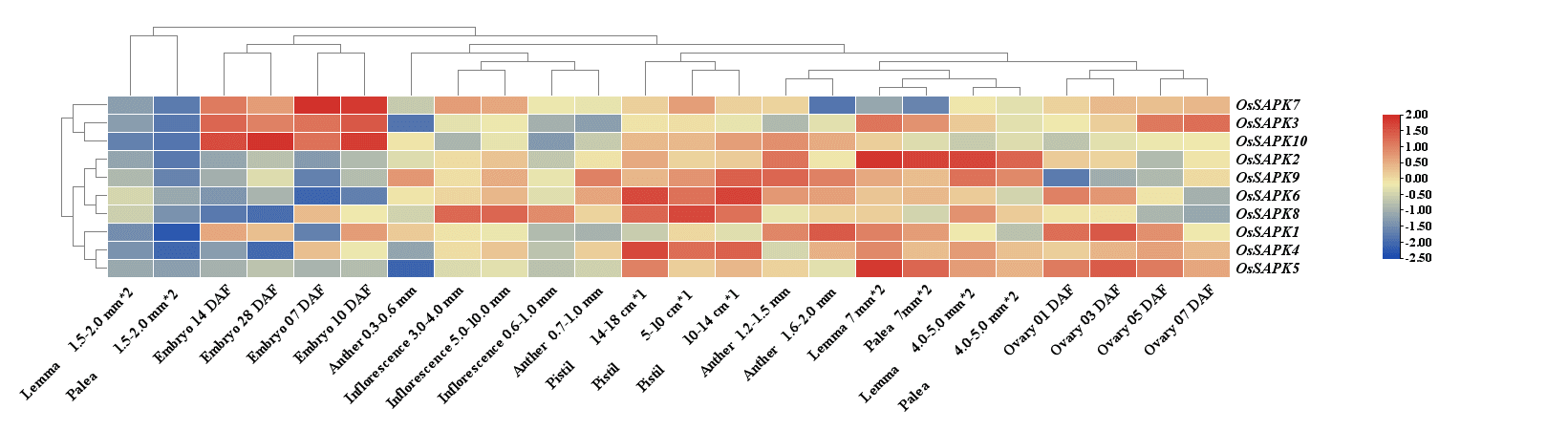
TBtools 热图美化可以参考:https://yuanj.top/posts/p5x7e7k5/
数据可用性
本文使用的所有数据均已保存至 GitHub:https://github.com/yuanj82/OsSnRK2-GFI