TBtools 快速完成基因功能注释
本文总阅读量 次
基因功能注释质量好坏取决于数据库质量高低,是否全面。于是,本地化进行基因功能注释,需要收集尽可能多的数据库(这个其实很不实际),也需要有较好的计算资源。通过使用网页服务工具,可以克服这个问题。我们可以一直使用最新最全的数据库,同时不需要消耗本地计算资源。
eggNOG-mapper 大名鼎鼎,是一款非常全面,高效,准确,且一直在更新的软件,对应的,该团队提供了网页接口,任何人可以提交蛋白序列文件,在极短的时间内(一般几分钟)完成基因功能注释,包括:具体功能描述信息、Gene Onotoloy 注释信息、KEGG 注释信息、PFAM 注释信息以及其他…
eggNOG-mapper 注释
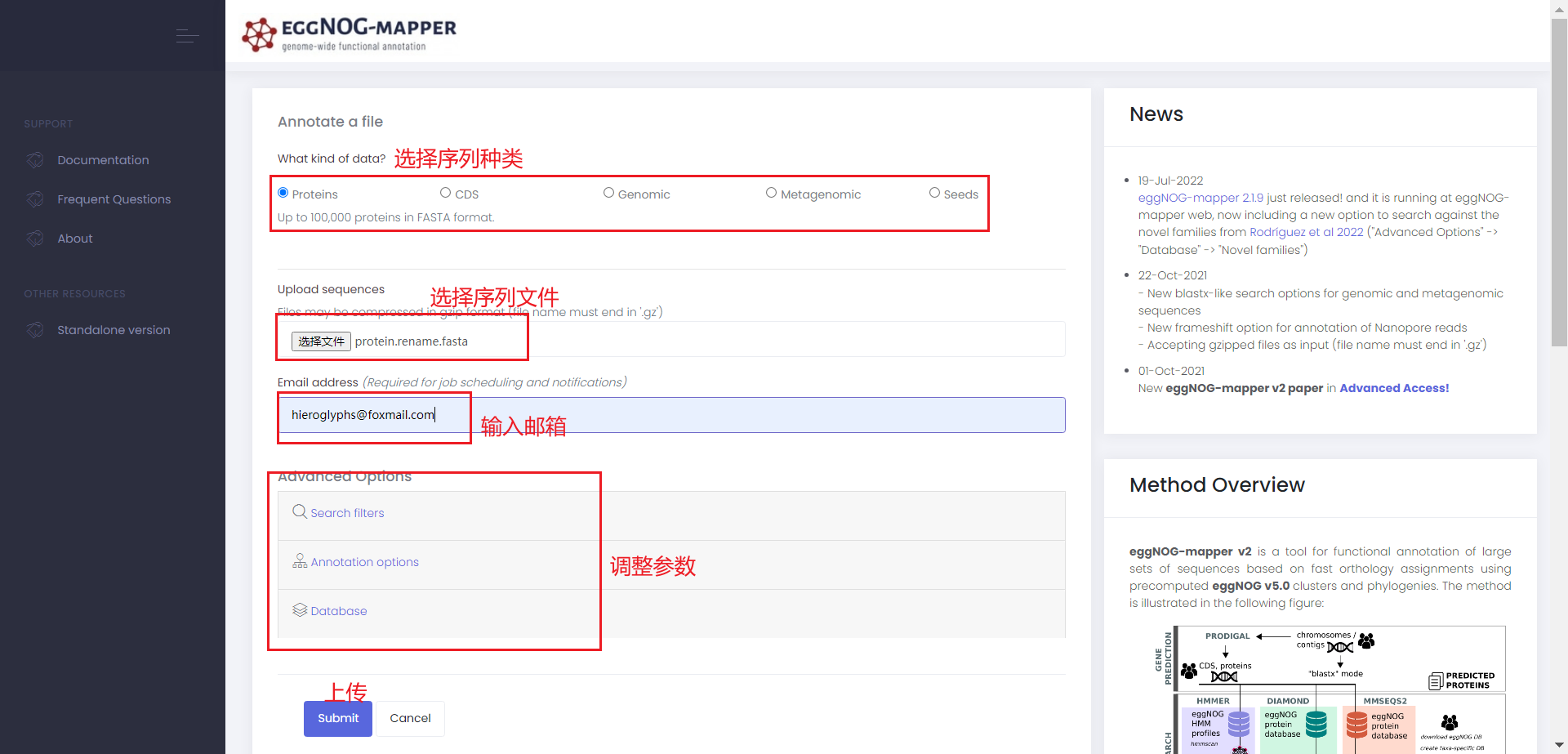
打开 eggNOG-mapper 主页 上传序列文件并输入邮箱,参数一般就使用默认即可,一般使用蛋白质序列
随后就可以看到右键提示
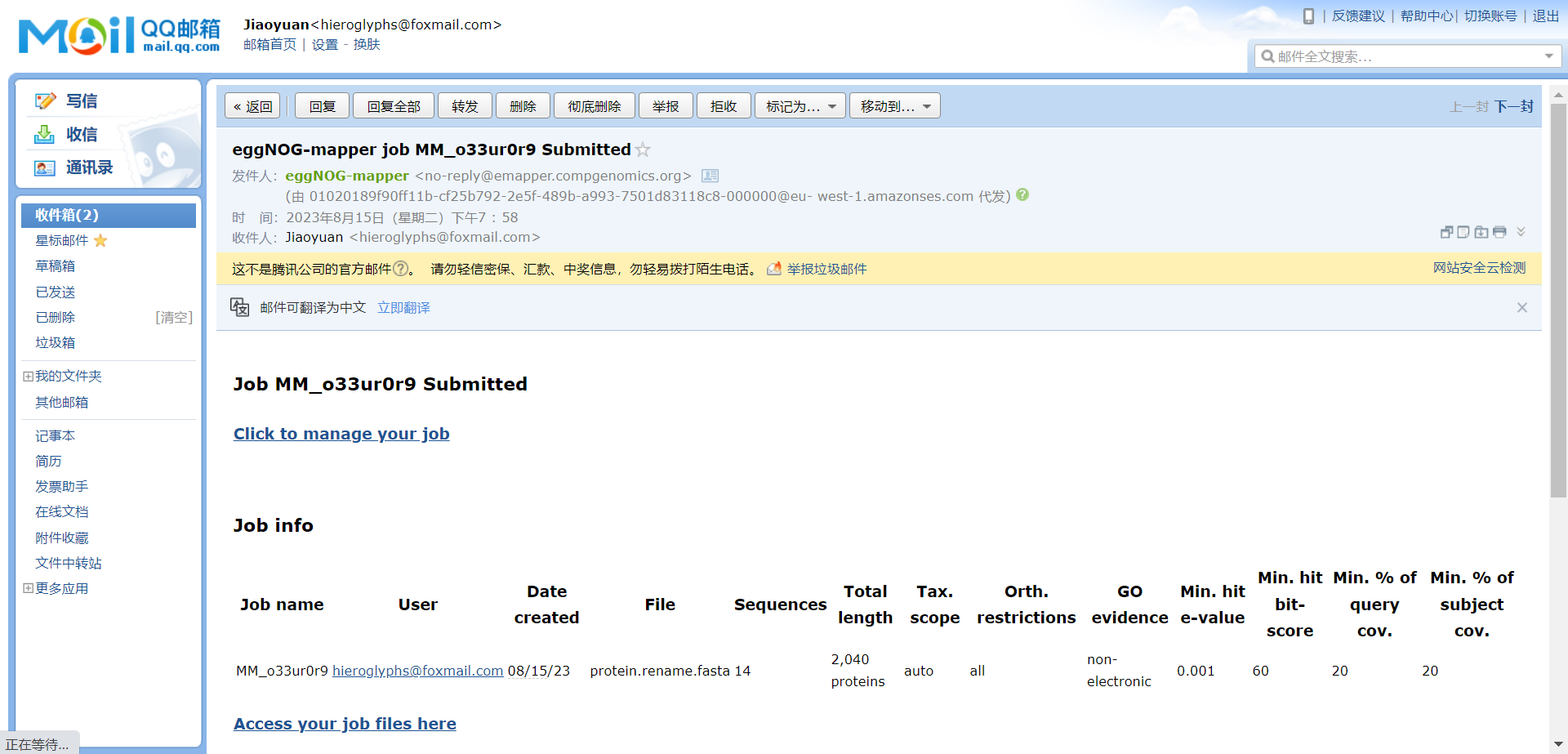
点击 Click to manage your job,启动工作

任务完成后,点击 Access your job files here 查看文件
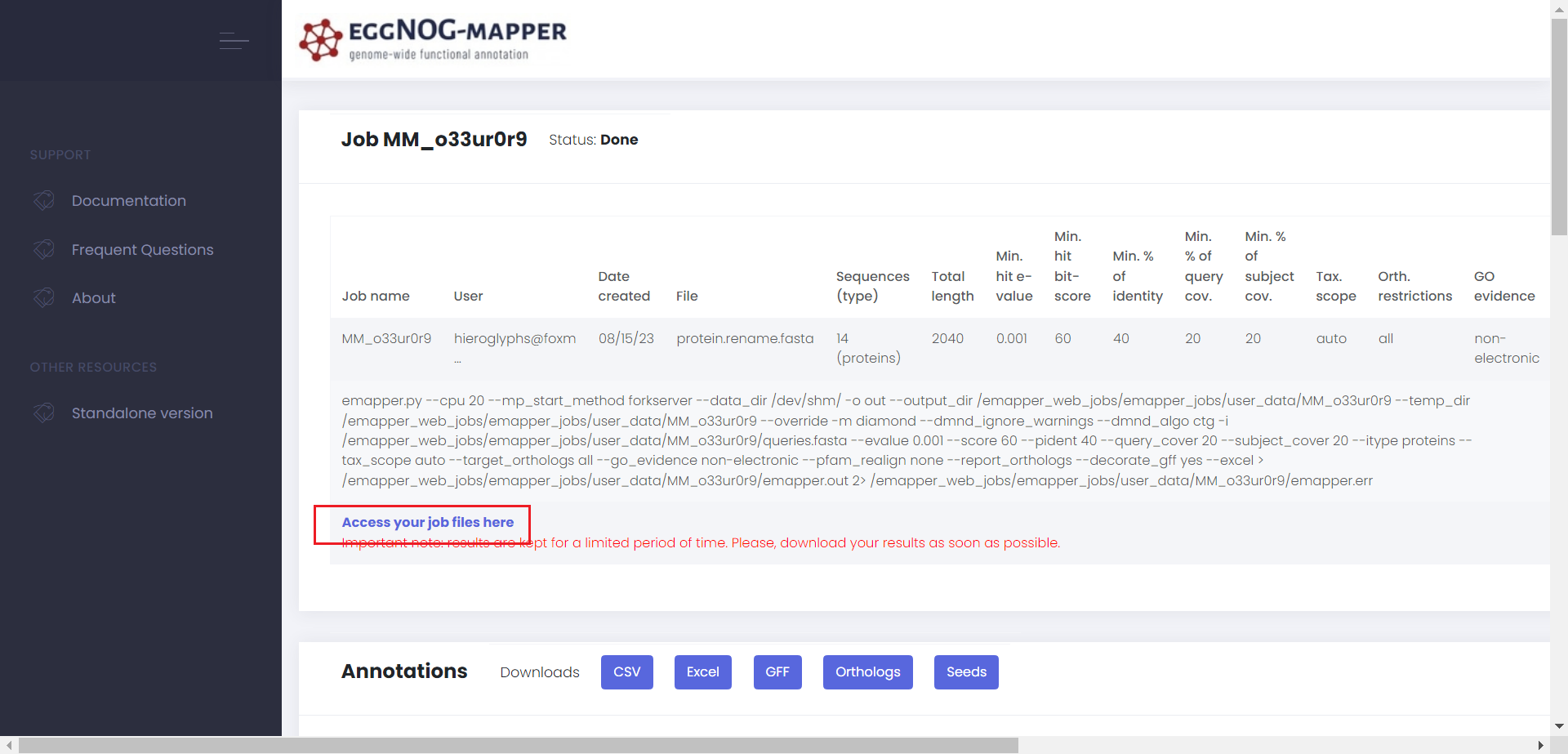
只需要下载 out.emapper.annotations 这个文件

可以用 Excel 打开文件查看信息
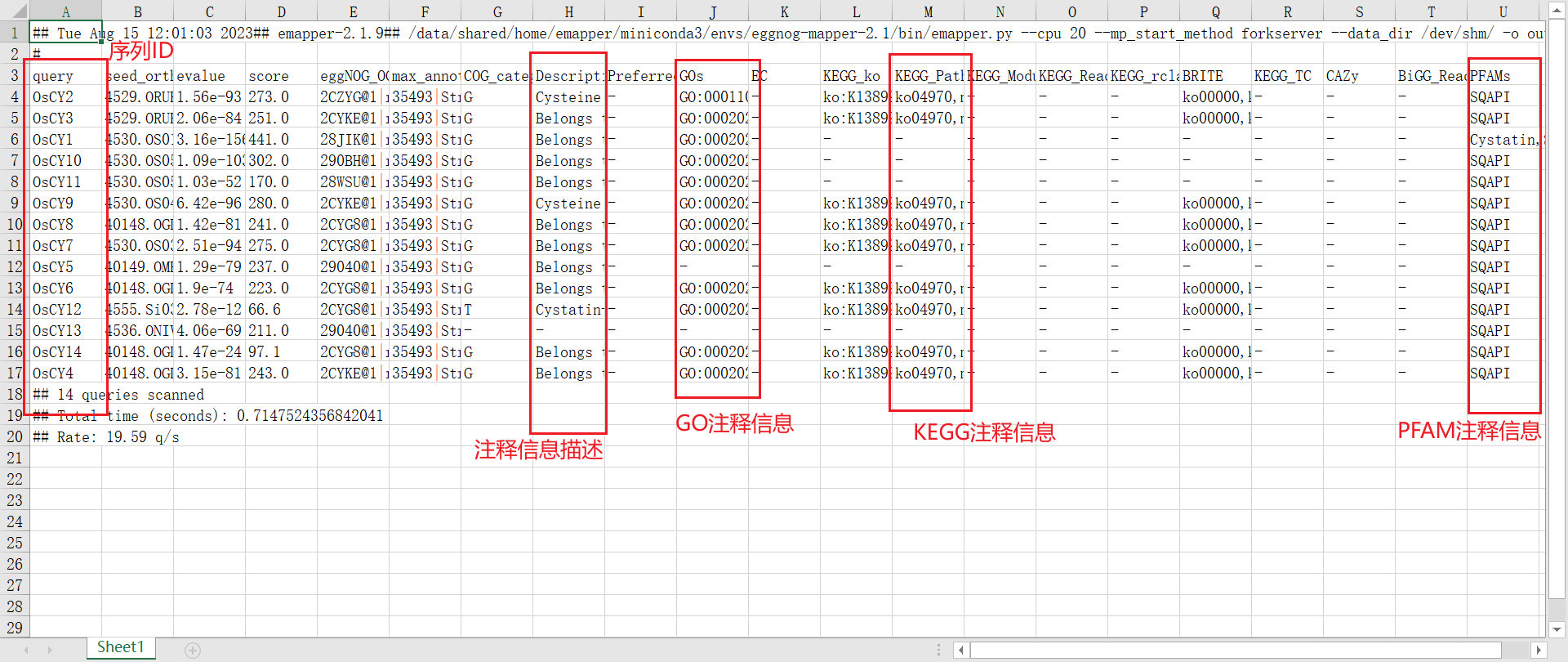
结果很全面,只是还是不能满足我们的需求
eggNOG-mapper Helper
接下来我们便使用 TBtools 的 eggNOG-mapper Helper 插件来整理结果
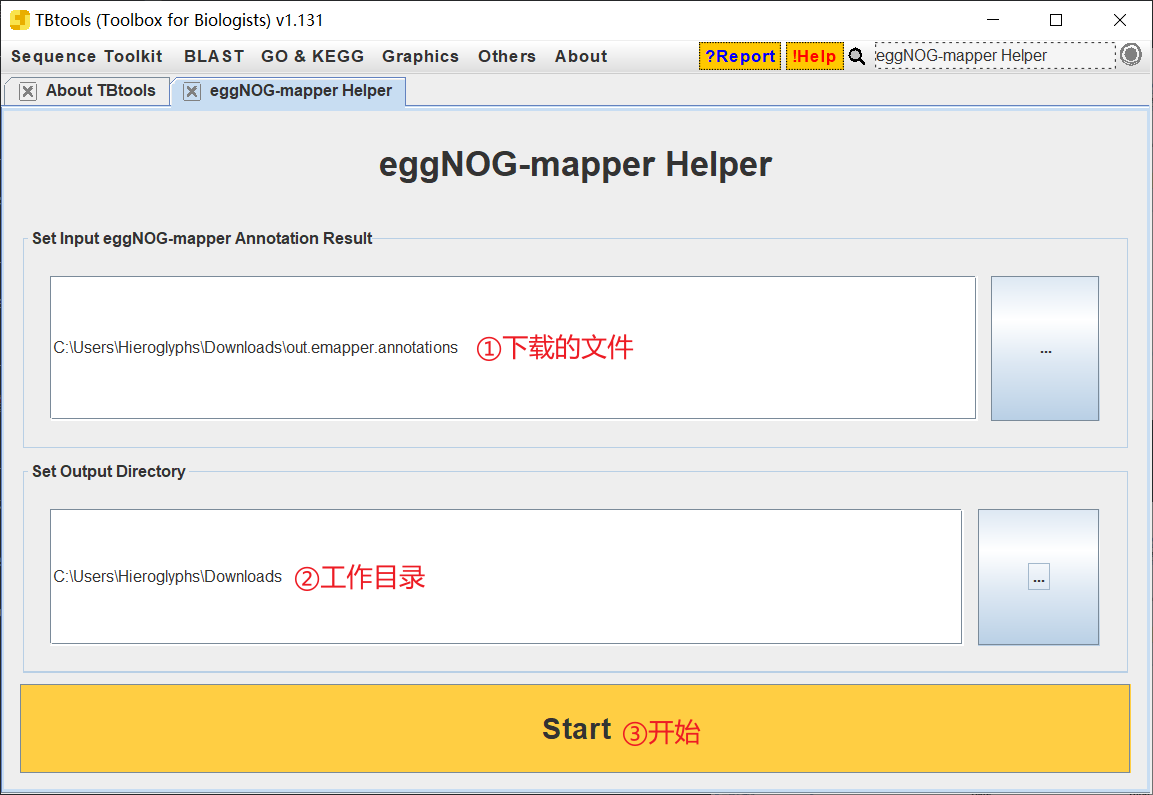
很快就能完成,完成后就可以查看整理好的信息

输出文件中,大家可能最关注的有四个:
- out.emapper.annotations.description.txt,对应的功能文本描述
- out.emapper.annotations.GO.txt,对应的是 GO 注释结果,可直接用于 TBtools GO 富集分析,当注释背景文件
- out.emapper.annotations.KEGG_Knum.txt,对应的是 KEGG 注释结果,可直接用于 TBtools KEGG 富集分析,当背景注释文件
- out.emapper.annotations.pfam.domain.txt,对应的是 PFAM 结构域注释,注意,这个注释结果是定性的,即有无某结构域,如果一个序列有多个相同结构域,只会显示一个