ATAC-Seq 分析流程
本文总阅读量 次
ATAC-Seq 是“Assay for Transposase-Accessible Chromatin with high-throughput Sequencing”的缩写。 ATAC-Seq 方法依赖于使用高活性转座酶 Tn5 的下一代测序(NGS)文库的构建。将 NGS 接头连接到转座酶上,该转座酶可以使染色质断裂并同时将这些接头整合到开放的染色质区域中。构建的文库可通过 NGS 测序,并使用生物信息学分析具有可及或可访问染色质的基因组区域。
ATAC-seq 概述
尽管 ATAC-seq 实验方法相对简单且稳定,但是专门为 ATAC-seq 测序数据开发的生物信息学分析软件却非常少。由于 ATAC-seq 和 ChIP-seq 数据的相似性较高,ChIP-seq 分析使用的软件一般也可用于 ATAC-seq 的分析,但是使用 ChIP-seq 软件分析得到的 ATAC-seq 结果尚未得到系统性的评估。

- step1:质控
- step2:比对
- step3:峰的召回
- step4:峰的注释
- step5:差异分析
- step6:基序分析、通路分析、核小体占据率分析、和其他组学分析的整合
上游的预处理部分与 RNA-seq 的差不多,可以参考我之前的文章:https://yuanj.top/posts/z6q5z7s5/
版本信息
- 成都理工大学超算平台 Red Hat 4.8.5-36
- conda 4.10.3
- R 4.4.0
- 测序数据来自 NCBI:PRJNA1080287
- 参考基因组和注释文件来自 Ensembl Plants
- 物种:Oryza sativa Japonica
一些名词解释
- peaks:峰。常用来表示染色质的开放程度,因为是测序的 reads 落在了染色质的开放区,堆叠后被可视化的一种丰度的体现。
- THSs:转座酶超敏感位点。
- CREs:顺式调控元件。即 DNA 分子中具有转录调节功能的特异 DNA 序列。按功能特性,真核基因顺式作用元件分为启动子、增强子及沉默子。
- ACRs:染色质开放区域。即正常或核小体被酶切裸露出来的 DNA 片段所在的区域。
- transposon:转座子。一段可以从原位上单独复制或断裂下来,环化后插入另一位点,并对其后的基因起调控作用的 DNA 序列。
- promoter:启动子。启动子是位于结构基因 5’端上游的 DNA 序列,能活化 RNA 聚合酶,使之与模板 DNA 准确的结合并具有转录起始的特异性。每个启动子包括至少一个转录起始点以及一个以上的功能组件。
- proximal promoters:近端启动子。是 DNA 上位于基因开始之前的一个区域,在那里蛋白质和其他分子结合在一起准备读取该基因。
- enhancer:增强子。增强子是远离转录起始点、决定基因的时间、空间特异性表达、增强启动子转录活性的 DNA 序列,其发挥作用的方式通常与方向、距离无关,可位于转录起始点的上游或下游。从功能上讲,没有增强子存在,启动子通常不能表现活性;没有启动子时,增强子也无法发挥作用。
- TFs:转录因子是保证目的基因以特定的强度在特定的时间与空间表达的蛋白质分子。与 RNA 聚合酶Ⅱ形成转录起始复合体,共同参与转录起始的过程。
- TSS:转录起始位点。在一个典型的基因内部,排列顺序为转录起始位点(TSS,一个碱基)-起始密码子编码序列-终止密码子编码序列-转录终止位点,即 TSS-ATG-TGA-TTS。
- histone:组蛋白。通常含有 H1,H2A,H2B,H3,H4 等 5 种成分,其中 H1 与 H3 极度富含赖氨酸,H1 不保守,其他组蛋白的基因非常保守。除 H1 外,其他 4 种组蛋白均分别以二聚体(共八聚体)相结合,形成核小体核心。DNA 便缠绕在核小体的核心上。而 H1 则与核小体间的 DNA 结合。
- nucleosome:核小体。是由 DNA 和组蛋白形成的染色质基本结构单位。每个核小体由 146bp 的 DNA 缠绕组蛋白八聚体 1.75 圈形成。核小体核心颗粒之间通过 50bp 左右的连接 DNA 相连,暴露在核小体表面的 DNA 能被特定的核酸酶接近并切割。
软件安装
只需要使用 conda 就可以安装所有需要的软件,主要使用的软件有以下一些:
- sra-tools:快速下载 NCBI SRA 数据
- fastQc:测序数据质量检测与控制
- multiqc:合并质量检测报告
- trim_galore:过滤低质量序列
- bwa:测序数据的回帖
- samtools:对 hisat2 比对的结果进行排序和压缩
- macs2:进行 Peak Calling
- deeptools:计算基因组区段 reads 覆盖度
先创建 conda 虚拟环境,安装所需要的软件,可以自行手动安装,也可以直接导入我的 conda 环境:
git clone https://github.com/yuanj82/NGS-analysis.git
cd NGS-analysis/environment
cp .condarc ~/
conda env create --file atac-seq-env.yml # ATAC-seq
创建完成后激活环境就可以使用了:
conda activate atac-seq
数据获取与预处理
从 NCBI SRA 数据库下载 SRR_Acc_List.txt 文件:
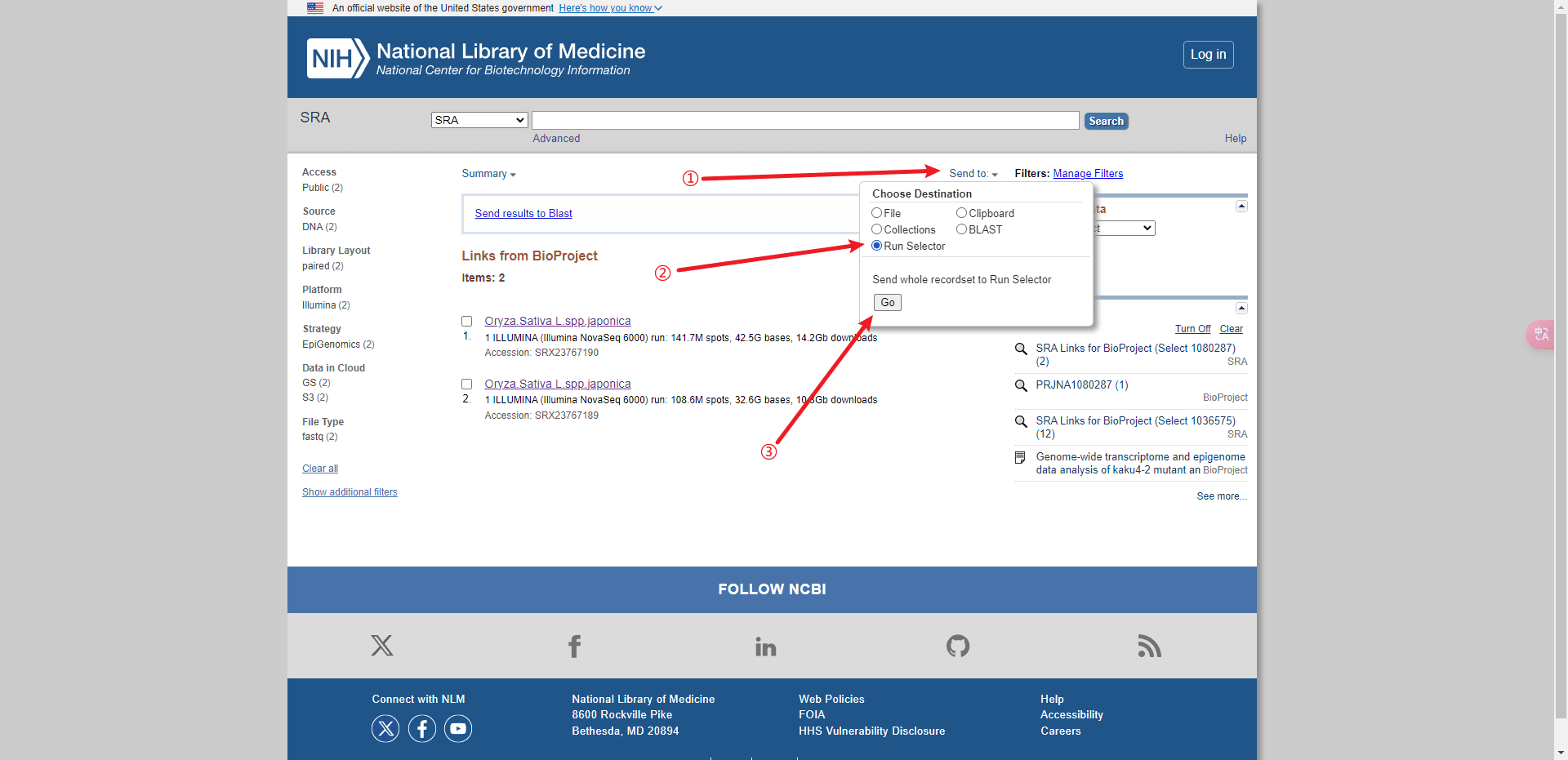
后面的分析都可以基于这个文件进行批处理。
使用 sratools 进行批量下载,下载比较慢,就用 nohup 挂在后台下载,自己可以做点别的事:
nohup prefetch -O . $(<SRR_Acc_List.txt) &
下载后的数据是放在一个个 SRRXXXXX 的文件夹里面的,我们用一个小脚本把它全部移动到一个 sra 文件夹便于处理:
mkdir sra
cat ./SRR_Acc_List.txt | while read id; do
mv -f "${id}/${id}.sra" ./sra/
rm -rf "${id}"
done
刚刚下载好的数据是 sra 格式的,使用 sratools 将其拆分并输出到 fastqgz 文件夹:
mkdir fastqgz
nohup fastq-dump --gzip --split-3 ./sra/*.sra --outdir ./fastqgz &
- –gzip 是将拆分的 fastq 文件压缩归档为 gz 格式
- –split-3 是将文件拆分为正向序列和逆向序列
参考基因组及注释文件
植物的我一般在 Ensembl Plants 下载,用 wget 或 curl 都可以,内存不大,这组数据是水稻的,所以就从 Ensembl Plants 下载水稻的基因组和注释文件:
wget https://ftp.ensemblgenomes.ebi.ac.uk/pub/plants/release-57/fasta/oryza_sativa/dna/Oryza_sativa.IRGSP-1.0.dna.toplevel.fa.gz
wget https://ftp.ensemblgenomes.ebi.ac.uk/pub/plants/release-57/gff3/oryza_sativa/Oryza_sativa.IRGSP-1.0.57.gff3.gz
然后解压:
gzip -d Oryza_sativa.IRGSP-1.0.57.gff3.gz
gzip -d Oryza_sativa.IRGSP-1.0.dna.toplevel.fa.gz
FastQc 质控
常用参数
fastqc [-o output dir] [–(no)extract] [-f fastq|bam|sam] [-c contaminant file] seqfile1 .. seqfileN
- -o –outdir:FastQC 生成的报告文件的储存路径,生成的报告的文件名是根据输入来定的
- –extract:生成的报告默认会打包成 1 个压缩文件,使用这个参数是让程序不打包
- -t –threads:选择程序运行的线程数,每个线程会占用 250MB 内存,越多越快咯
- -c –contaminants:污染物选项,输入的是一个文件,格式是 Name [Tab] Sequence,里面是可能的污染序列,如果有这个选项,FastQC 会在计算时候评估污染的情况,并在统计的时候进行分析,一般用不到
- -a –adapters:也是输入一个文件,文件的格式 Name [Tab] Sequence,储存的是测序的 adpater 序列信息,如果不输入,目前版本的 FastQC 就按照通用引物来评估序列时候有 adapter 的残留
- -q –quiet:安静运行模式,一般不选这个选项的时候,程序会实时报告运行的状况
进行质量检测
直接进行批处理:
fastqc ./fastqgz/*.fastq.gz -o ./fastqc_report
当然,数据比较多的时候还是挂在后台批处理然后等着就行:
nohup fastqc SRR*.fastq.gz &
multiqc ./fastqc_report # 将多个质量检测报告合并
质量检测报告查看和解读之前的文章有,这里就不赘述了。
Trim Galore 过滤低质量序列
常用参数
trim_galore [options] <filename(s)>
- –quality:设定 Phred quality score 阈值,默认为 20
- –phred33:选择-phred33 或者-phred64,表示测序平台使用的 Phred quality score
- –adapter:输入 adapter 序列。也可以不输入,Trim Galore! 会自动寻找可能性最高的平台对应的 adapter。自动搜选的平台三个,也直接显式输入这三种平台,即–illumina、–nextera 和–small_rna
- –stringency:设定可以忍受的前后 adapter 重叠的碱基数,默认为 1(非常苛刻),可以适度放宽,因为后一个 adapter 几乎不可能被测序仪读到
- –length:设定输出 reads 长度阈值,小于设定值会被抛弃
- –paired:对于双端测序结果,一对 reads 中,如果有一个被剔除,那么另一个会被同样抛弃,而不管是否达到标准
- –retain_unpaired:对于双端测序结果,一对 reads 中,如果一个 read 达到标准,但是对应的另一个要被抛弃,达到标准的 read 会被单独保存为一个文件
- –gzip 和–dont_gzip:清洗后的数据 zip 打包或者不打包
- –output_dir:输入目录。需要提前建立目录,否则运行会报错
- – trim-n :移除 read 一端的 reads
过滤低质量序列
使用一个批处理对所有数据进行处理:
mkdir clean
cat ./SRR_Acc_List.txt | while read id; do
trim_galore \
-q 20 \
--length 36 \
--max_n 3 \
--stringency 3 \
--fastqc \
--paired \
-o ./clean/ \
./fastqgz/${id}_1.fastq.gz ./fastqgz/${id}_2.fastq.gz
done
完成后文件会输出到 clean 文件夹。
bwa 回帖到参考基因组
常用参数
bwa index [ –p prefix ] [ –a algoType ] <in.db.fasta>
- -P STR:输出数据库的前缀;默认和输入的文件名一致,输出的数据库在其输入文件所在的文件夹,并以该文件名为前缀
- -a [is|bwtsw]:构建 index 的算法,有两个算法:is 是默认的算法,虽然相对较快,但是需要较大的内存,当构建的数据库大于 2GB 的时候就不能正常工作了;bwtsw 对于短的参考序列式不工作的,必须要大于等于 10MB, 但能用于较大的基因组数据,比如人的全基因组
bwa mem [options] ref.fa reads.fq [mates.fq]
- -t INT:线程数,默认是 1
- -M:将 shorter split hits 标记为次优,以兼容 Picard’s markDuplicates 软件
- -p:若无此参数:输入文件只有 1 个,则进行单端比对;若输入文件有 2 个,则作为 paired reads 进行比对。若加入此参数:则仅以第 1 个文件作为输入(输入的文件若有 2 个,则忽略之),该文件必须是 read1.fq 和 read2.fa 进行 reads 交叉的数据
- -R STR:完整的 read group 的头部,可以用 ‘\t’ 作为分隔符, 在输出的 SAM 文件中被解释为制表符 TAB. read group 的 ID,会被添加到输出文件的每一个 read 的头部
- -T INT:当比对的分值比 INT 小时,不输出该比对结果,这个参数只影响输出的结果,不影响比对的过程
- -a:将所有的比对结果都输出,包括 single-end 和 unpaired paired-end 的 reads,但是这些比对的结果会被标记为次优
进行比对
使用前面下载的水稻基因组建立索引:
bwa index -a bwtsw Oryza_sativa.IRGSP-1.0.dna.toplevel.fa Oryza_sativa
批量进行比对:
cat ./SRR_Acc_List.txt | while read id;
do
bwa mem -v 3 -t 4 ./Oryza_sativa/Oryza_sativa.IRGSP-1.0.dna.toplevel.fa ./clean/${id}_1.fastq.gz ./clean/${id}_2.fastq.gz -o ./compared/${id}.sam
done
比对后的结果会输出到 compared 文件夹。
samtools 排序压缩
这个就很常见了,几乎是组学分析必用的软件,之前也详细介绍过了,不再赘述,使用下列批处理将所有数据排序压缩并输出到 sorted 文件夹:
cat ./SRR_Acc_List.txt | while read id ;do
samtools \
sort \
-n -@ 5 \
./compared/${id}.sam \
-o ./sorted/${id}.bam
done
接着需要给每个 bam 文件创建索引,后面会用到:
cat ./SRR_Acc_List.txt | while read id;
do
samtools index \
-@ 4 \
./sorted/${id}.bam
done
macs2 进行 Peak Calling
这是非常关键的一步,ATAC-seq 分析的核心结果都要从这里出,使用的是 macs2。
常用参数
macs2 [-h] [--version] {callpeak,bdgpeakcall,bdgbroadcall,bdgcmp,bdgopt,cmbreps,bdgdiff,filterdup,predictd,pileup,randsample,refinepeak} ...
- -f 指定输入文件的格式,如 SAM、BAM、BED 等
- -c 对照组,可以接多个数据,空格分隔
- -t 实验组,ChIP-seq 数据,可以接多个数据,空格分隔
- -n 输出文件名的前缀
- -g 有效基因组大小,软件有几个默认值,如 hs 指 human
- –outdir 输出结果的存储路径
- –bdg 输出 bedgraph 格式的文件
- -q FDR 阈值,默认为 0.05
Peak Calling
水稻基因组大小是 3.6e+8,这个基因组的大小是很关键的,如果指定错误或者未指定都会影响后面的分析。使用下列批处理进行分析:
cat ./SRR_Acc_List.txt | while read id;do
macs2 \
callpeak \
-t ./sorted/${id}.bam \
-f BAM \
-g 3.6e+8 \
-n ./peaks/${id} \
done
完成后每个样本会输出几个文件:
- NAME_model.r:可视化双峰模型的 R 代码,对双端测序而言,它本身测的就是文库的两端,因此不用建立模型和偏倚,我们只需要对 MACS 设置参数 –nodel 就能略过双峰模型建立的过程
- NAME_peaks.narrowPeak:BED 6+4 格式
- NAME_peaks.xls:包含 peak 信息的 xls 文件,注意 chrStart 是 1-based
- NAME_peaks_summits.bed:BED 格式,包含 peak 的 summits 位置,第 5 列是-log10pvalue,如果想找 motif,推荐使用此文件
deeptools 计算基因组区段 reads 覆盖度
deeptools 包含了很多的工具:
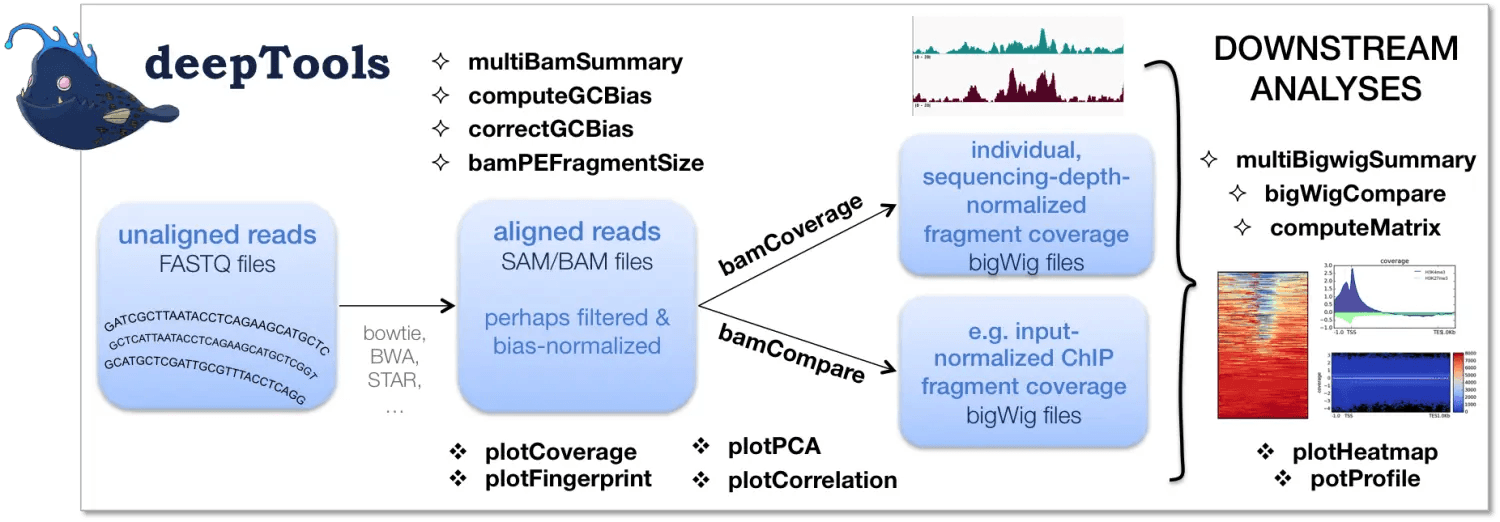
这里我们主要使用的是 bamCoverage 和 computeMatrix。
bamCoverage
bamCoverage 利用测序数据比对结果转换为基因组区域 reads 覆盖度结果。可以自行设定覆盖度计算的窗口大小 (bin)。
bamCoverage -b reads.bam -o coverage.bw

必须的参数:
- –bam, -b:要处理的 BAM 文件
- –outFileName, -o:输出文件名
- –outFileFormat, -of:输出文件的格式,可以是 bigwig、bedgraph
标准化参数:
- –normalizeUsing:选择:RPKM,CPM,BPM,RPGC,None
- RPKM :每千碱基每百万映射 reads 数,RPKM (per bin) = number of reads per bin / (number of mapped reads (in millions) * bin length (kb))
- CPM :每百万映射 reads 数;, CPM (per bin) = number of reads per bin / number of mapped reads (in millions)
- BPM :每百万映射 reads 数的 bin,与 RNA-seq 中的 TPM 相同;BPM (per bin) = number of reads per bin / sum of all reads per bin (in millions).
- RPGC :基因组内容的 reads 数(1x 归一化), number of reads per bin / scaling factor for 1x average coverage.
- None=默认值,相当于根本不设置此选项
可选的参数很多,可以参考 deeptools 的手册。
此处我使用的命令是:
cat ./SRR_Acc_List.txt | while read id;do
bamCoverage \
-b ./sorted/${id}.bam \
-o ./bigwig/${id}.bw \
--normalizeUsing RPKM
done
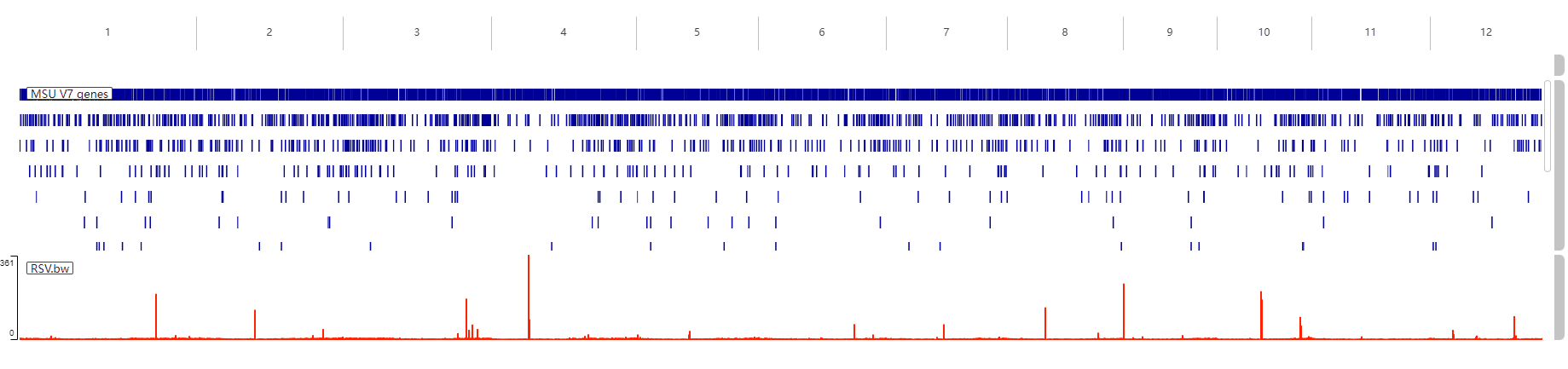
这里就需要我们前面给 bam 文件创建的 samtools 索引,不然的话会报错。完成之后会输出一系列 bw 文件,这个文件可以导入到 IGV 进行可视化:
computeMatrix
computeMatrix [-h] [--version] ...
- reference-point:单个输入文件模式
- scale-regions:多个输入文件模式
必须的参数:
- –regionsFileName, -R:文件名或名称,采用 BED 或 GTF 格式,包含要绘制的区域,如果给出多个床文件,则每个床文件都被视为可以单独绘制的组
- –scoreFileName, -S:bigWig 文件包含要绘制的分数,多个文件应以空格分隔,BigWig 文件可以使用 bamCoverage 或 bamCompare 工具获取
输出参数:
- –outFileName, -out, -o:用于保存“plotHeatmap”和“plotProfile”工具所需的 gzip 压缩矩阵文件的文件名
- –outFileNameMatrix:指定热图矩阵的名称
- –outFileSortedRegions:跳过零或最小/最大阈值后保存区域的文件名,文件中区域的顺序遵循所选的排序顺序
此处我对单个文件进行批处理计算:
cat ./SRR_Acc_List.txt | while read id;do
computeMatrix reference-point \
--referencePoint TSS \
--missingDataAsZero \
-b 3000 -a 3000 \
-R ${id}.bed \
-S ${id}.bw \
-o ${id}.TSS.gz
done
- –referencePoint TSS:指定参考点为转录起始位点 (TSS)
- –missingDataAsZero:如果数据缺失,则将其视为零处理
- -b 3000 -a 3000:定义参考点附近的区域大小,这里设置为上游和下游各 3000 个碱基
- -R:指定参考文件,${id} 是当前循环中的 ID
- -S:指定测序数据文件,${id}.bw 是当前循环中的 ID 对应的 bigWig 格式的文件
- -o:指定输出文件名,${id}.TSS.gz 是当前循环中的 ID 作为前缀的输出文件名
绘制热图与折线图:
cat ./SRR_Acc_List.txt | while read id;do
plotHeatmap -m ${id}.TSS.gz -out ${id}.TSS.png
done
再同时对多个样本进行计算,将输出结果放在一个文件:
computeMatrix reference-point \
--referencePoint TSS \
--missingDataAsZero \
-b 3000 -a 3000 \
-R SRR28122456.bed \
-S SRR28122455.bw SRR28122456.bw \
-o TSS.gz
计算完成之后绘制多个样本的折线图在同一个图内:
plotProfile --dpi 720 -m TSS.gz -out TSS.pdf --plotFileFormat pdf --perGroup

这个图左边是前面绘制的两个热图+折线图,右边是前面绘制多个样本折线图在同一图内。
Chipseek 可视化 peak
R 包 ChIPseeker 主要是为了能对 ChIP-seq 数据进行注释与可视化,主要对 peak 位置及 peak 邻近基因的注释。然而,在之后对 ChIPseeker 的应用中,发现它不局限于 ChIP-seq,可用于其他的 peak(如 ATAC-seq,DNase-seq 等富集得到的)注释,甚至还可用于 long intergenic non-coding RNAs (lincRNAs) 的注释。该包功能强大之处还是在于可视化上。
安装 ChIPseeker 包:
if (!require("BiocManager", quietly = TRUE))
install.packages("BiocManager")
BiocManager::install("ChIPseeker")
所需的文件有两个,一个是 BED 格式的文件,至少得有染色体名字、染色体起始位点和染色体终止位点,其它信息如 name,score,strand 等可有可无。这里直接用前面 macs2 输出的 bed 文件即可。另一个是有注释信息的 TxDb 对象,Bioconductor 包提供了 30 个 TxDb 包,包含了很多物种,如人,老鼠等。当所研究的物种没有已有的 TxDb 时,可通过 GenomicFeatures 包使用基因组注释文件进行制作:
if (!require("BiocManager", quietly = TRUE))
install.packages("BiocManager")
BiocManager::install("GenomicFeatures")
BiocManager::install("txdbmaker")
这里我使用前面下载的水稻注释文件进行制作:
library("GenomicFeatures")
spombe <- makeTxDbFromGFF("Oryza_sativa.IRGSP-1.0.57.gff3")
然后就可以读入 bed 文件进行可视化:
# 为了这里方便分析,将两个 bed 文件按照样本信息进行重命名了
Mock <- readPeakFile('./peaks/Mock.bed')
RSV <- readPeakFile('./peaks/RSV.bed')
Mock_peakAnno <- annotatePeak(Mock, tssRegion =c(-3000, 3000), TxDb = spombe)
RSV_peakAnno <- annotatePeak(RSV, tssRegion =c(-3000, 3000), TxDb = spombe)
plotAnnoPie(Mock_peakAnno)
plotAnnoPie(RSV_peakAnno)
也可以将多个样本绘制在同一个图:
peaks <- list(Mock_peakAnno=Mock, RSV_peakAnno=RSV)
peakAnno <- lapply(peaks, annotatePeak, tssRegion = c(-3000, 3000), TxDb = spombe)
plotAnnoBar(peakAnno)

在染色体上可视化 peaks 的位置
这个使用 TBtools 来进行,先制作一个染色体长度文件:
1 43270923
2 35937250
3 36413819
4 35502694
5 29958434
6 31248787
7 29697621
8 28443022
9 23012720
10 23207287
11 29021106
12 27531856
然后使用 TBtools 的 circos 绘图工具,只把染色体长度文件导入:
修改 macs2 输出文件中的 bed 文件,只保留染色体、开始位置、终止位置和值四列即可,如:
1 1074 1075 33.7137
1 84253 84254 2.21489
1 104155 104156 5.22729
1 104518 104519 3.93076
1 104876 104877 6.8855
将文件导入到 TBtools:

然后改一下颜色、字体什么的进行一下简单的美化:

到这里大致的流程基本上就完成了,还有一些分析就是视情况才做的了。